Anslået tid: 90 minutter
læringsmål:
- Beskrive processen af RNA-seq bibliotek forberedelse
- Beskrive Illumina sekventering metode
Introduktion til RNA-seq
RNA-seq er en spændende eksperimenterende teknik, der er anvendt til at undersøge og/eller kvantificere genekspression i eller mellem betingelser.,
som vi ved, giver gener instruktioner til fremstilling af proteiner, som udfører en funktion i cellen. Selvom alle celler indeholder den samme DNA-sekvens, er muskelceller forskellige fra nerveceller og andre typer celler på grund af de forskellige gener, der tændes i disse celler, og de forskellige RNA ‘ er og proteiner, der produceres.
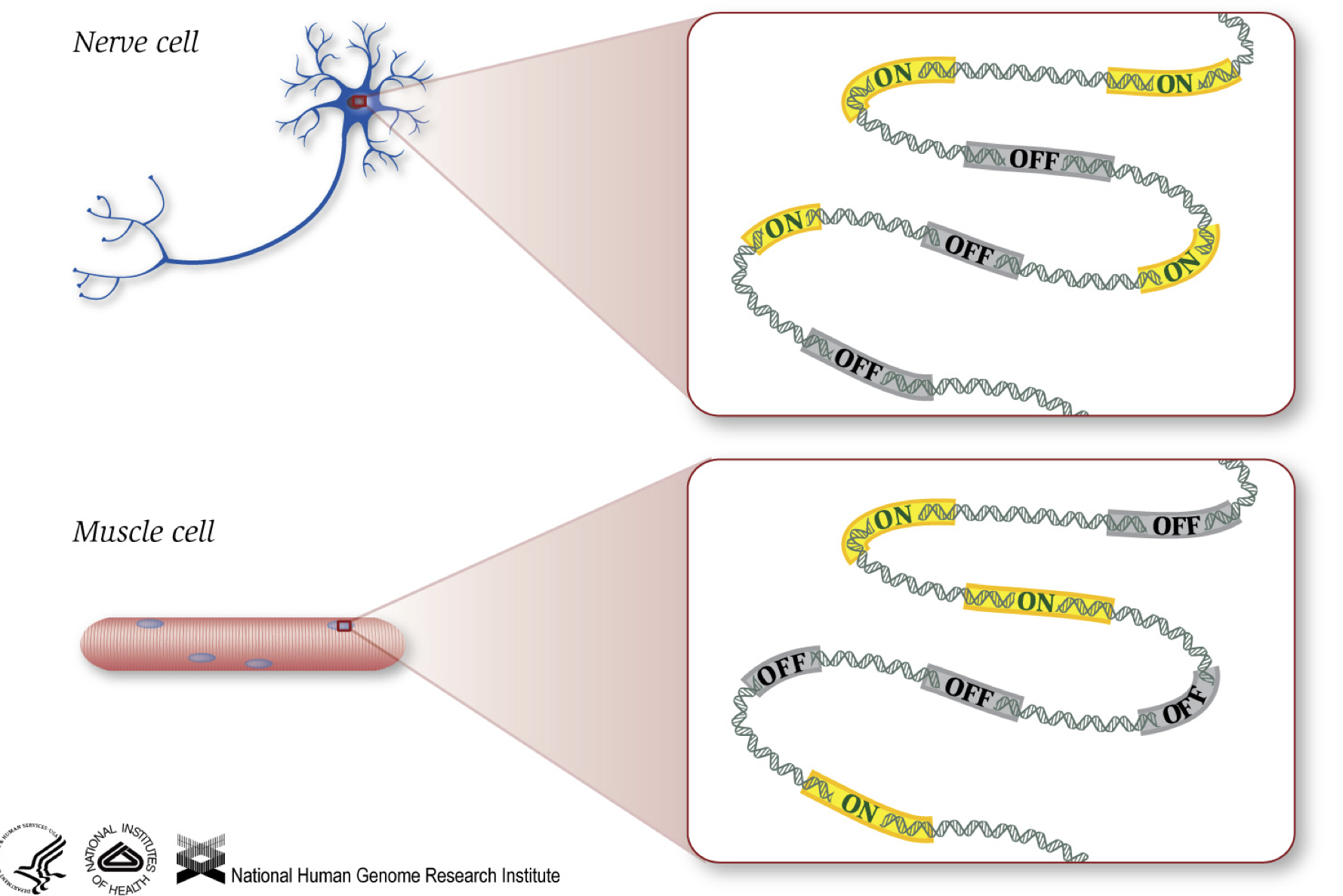
forskellige biologiske processer såvel som mutationer kan påvirke, hvilke gener der er tændt, og hvilke der er slukket, ud over, hvor meget specifikke gener der tændes / slukkes.,
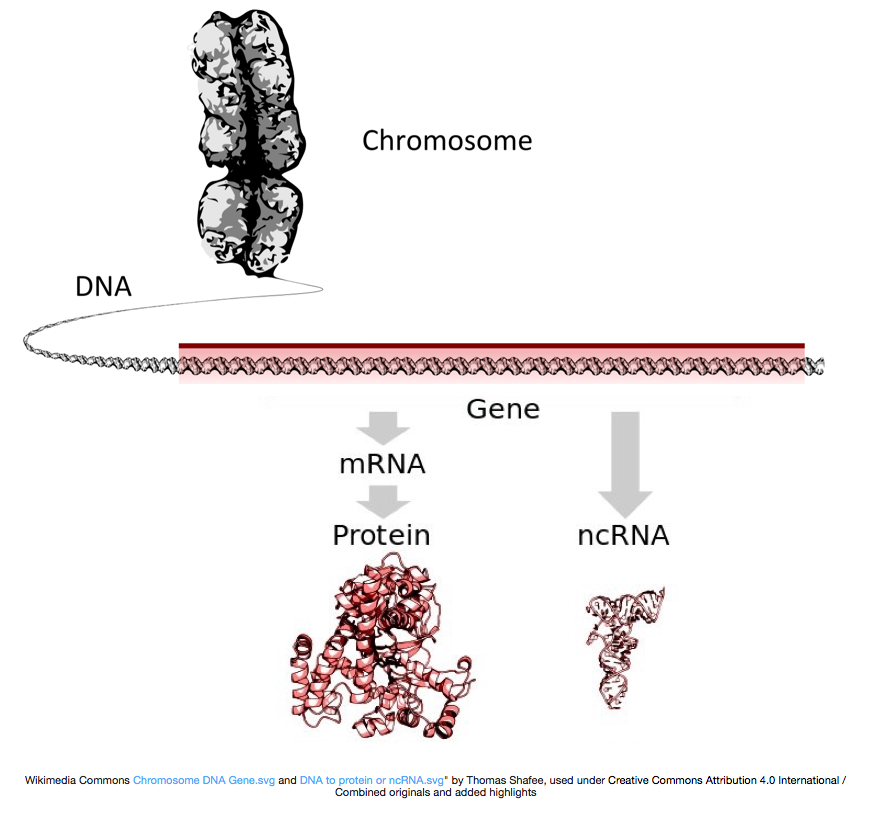
for at fremstille proteiner transkriberes DNA ‘ et til messenger RNA eller mRNA, som oversættes af ribosomet til protein. Nogle gener koder imidlertid RNA, der ikke bliver oversat til protein; disse RNA ‘er kaldes ikke-kodende RNA’ er eller ncrna ‘ er. Ofte har disse RNA ‘er en funktion i sig selv og omfatter rRNA’ er, tRNA ‘er og sirna’ er, blandt andre. Alle RNA ‘ er transkriberet fra gener kaldes transkripter.
for at blive oversat til proteiner skal RNA undergå behandling for at generere mRNA., I figuren nedenfor repræsenterer den øverste streng i billedet et gen i DNA ‘ et, der består af de uoversatte regioner (UTRs) og den åbne læseramme. Gener transkriberes til præ-mRNA, som stadig indeholder de introniske sekvenser. Efter post-transciptional behandling, intronerne splejses ud, og en polyA hale og 5’ cap tilsættes for at give modne mRNA-transkripter, som kan oversættes til proteiner.,
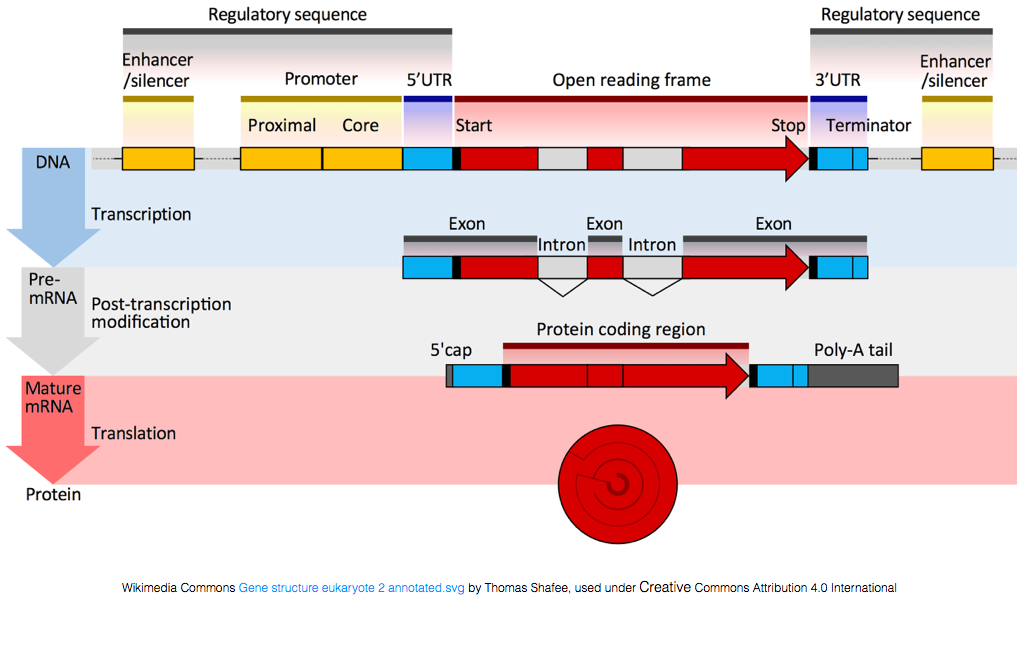
Mens mRNA afskrifter har en polyA hale, mange af de ikke-kodende RNA afskrifter ikke som post-transkriptionel behandlingen er forskellig for disse udskrifter.
Transkriptomics
transkriptomet er defineret som en samling af alle transkriptudlæsninger, der findes i en celle., RNA-seq data, der kan bruges til at udforske og/eller kvantificere transkriptom af en organisme, som kan blive brugt til følgende typer af eksperimenter:
- Differentiel Gen-Ekspression: kvantitativ evaluering og sammenligning af udskrift niveauer
- Transkriptom montering: bygning profil transskriberet regioner af genomet, en kvalitativ evaluering.,
- Kan bruges til at hjælpe med at opbygge bedre gen modeller, og kontrollere dem ved hjælp af forsamlingen
- Metatranscriptomics eller fællesskabet transkriptom-analyse
Illumina bibliotek forberedelse
Når du starter en RNA-seq eksperiment, for hver prøve RNA behov for at blive isoleret og forvandlet til et cDNA bibliotek for sekventering. Den generelle arbejdsgang til biblioteksforberedelse er detaljeret i de trinvise billeder nedenfor.
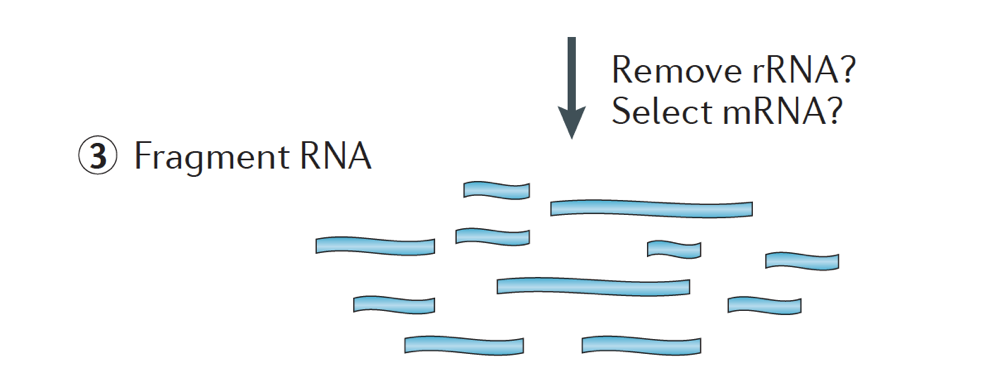
kort sagt isoleres RNA fra prøven, og kontaminerende DNA fjernes med DNase.,
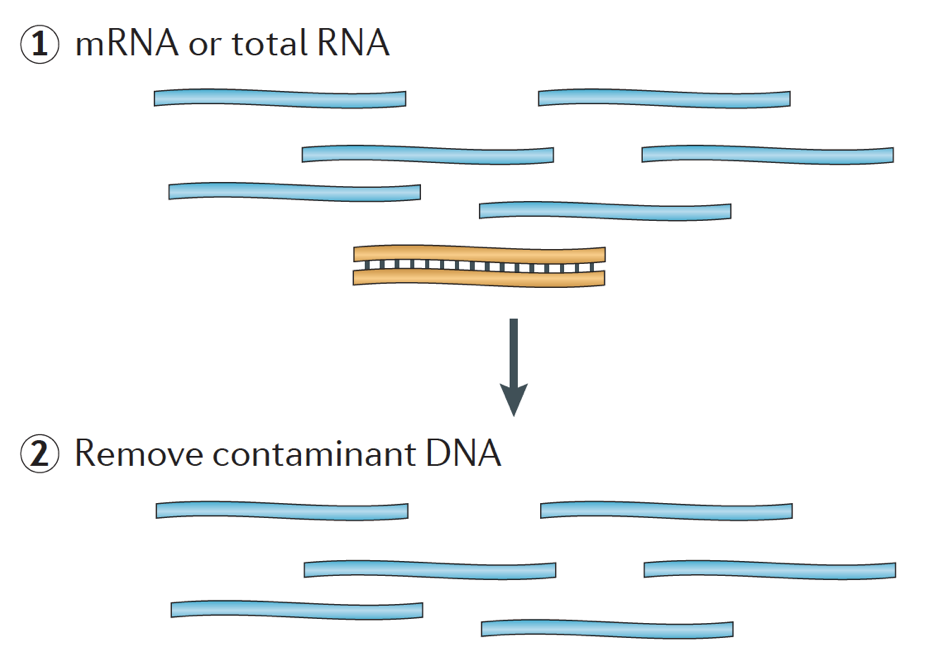
RNA-prøven gennemgår derefter enten valg af mRNA (polyA-udvælgelse) eller udtømning af rRNA. Det resulterende RNA er fragmenteret.
generelt repræsenterer ribosomalt RNA størstedelen af de RNA ‘er, der er til stede i en celle, mens messenger RNA’ er repræsenterer en lille procentdel af total RNA, ~2% hos mennesker. Derfor, hvis vi ønsker at studere de proteinkodende gener, er vi nødt til at berige for mRNA eller nedbryde rRNA., Til differentiel genekspressionsanalyse er det bedst at berige for Poly (a)+, medmindre du sigter mod at få information om lange ikke-kodende RNA ‘ er, så lav en ribosomal RNA-udtømning.
størrelsen af målfragmenterne i det endelige bibliotek er en nøgleparameter til bibliotekskonstruktion. DNA fragmentering sker typisk ved fysiske metoder (dvs, akustisk skæring og sonikering) eller enzymatiske metoder (dvs, ikke-specifikke endonuclease cocktails og transposase tagmentation reaktioner.,
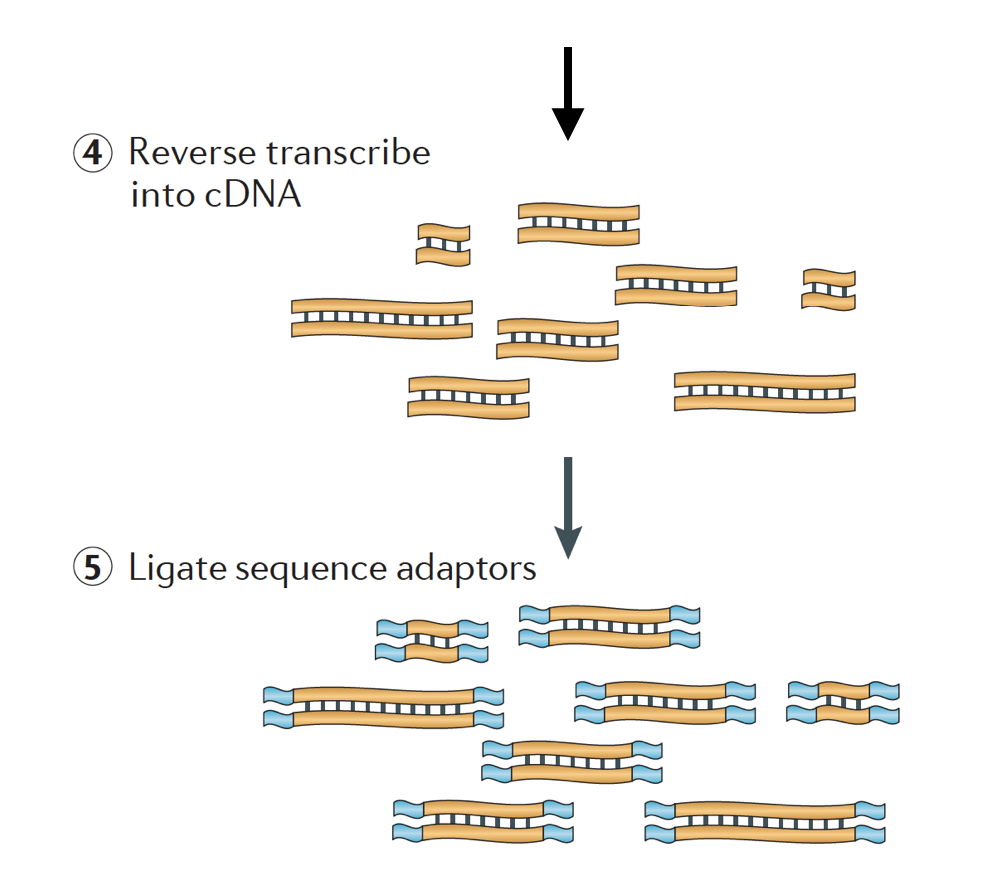
RNA ‘ et transkriberes derefter omvendt til dobbeltstrenget cDNA, og sekvensadaptere tilføjes derefter til enderne af fragmenterne.
cDNA-bibliotekerne kan genereres på en måde, så man kan bevare information om, hvilken DNA-streng RNA ‘ et blev transkriberet fra. Biblioteker, der bevarer disse oplysninger kaldes strandede biblioteker, som nu er standard med Illuminas Truse.strandede RNA-se. kits., Strandede biblioteker bør ikke være dyrere end unstranded, så der er ikke rigtig nogen grund til ikke at erhverve disse yderligere oplysninger.,
Der er 3 typer af cDNA-biblioteker:
- Videresend (secondstrand) – læser ligner gen-sekvens eller secondstrand cDNA sekvensen
- Reverse (firststrand) – læser ligner supplement af genet, sekvensen, eller firststrand cDNA sekvensen (TruSeq)
- Unstranded
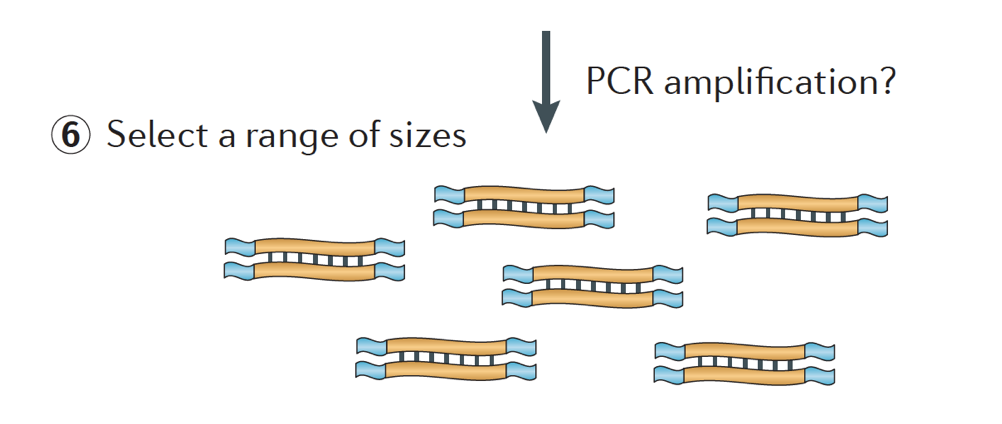
Endelig, andre fragmenter er så PCR-amplificeret hvis det er nødvendigt, og fragmenterne er størrelsen er valgt (normalt ~300-500bp) for at afslutte bibliotek.
Billede kredit: Martin J. A. Wang Z., Nat. Rev., Genet. (2011) 12:671-682
Illumina Sekventering
Single-end versus Parret-end
Efter udarbejdelsen af de biblioteker, sekventering kan udføres for at generere nukleotidsekvenser af enderne af de fragmenter, som kaldes læser. Du har valget mellem at sekventere en enkelt ende af cDNA-fragmenterne (single-end læser) eller begge ender af fragmenterne (parret-end læser).,
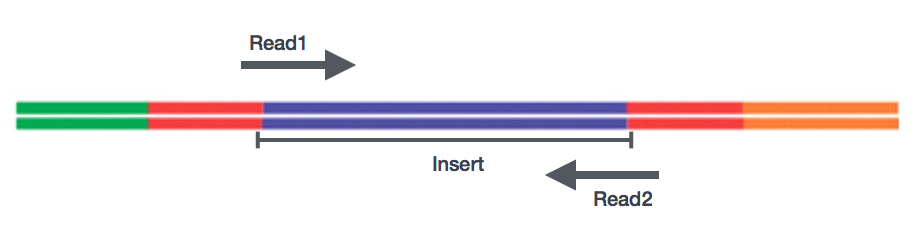
- SE – Single end datasæt => Kun Read1
- PE – Parret-end datasæt => Read1 + Read2
- kan være 2 separate FastQ filer, eller bare en med interleaved par
Generelt single-end-sekventering er tilstrækkeligt, hvis det forventes, at læser vil matche flere steder på genomet (fx organismer med mange paralogous gener), samlinger er udført, eller for at splejse isoform differentiering. Vær opmærksom på, at parret-ende læser er generelt 2x dyrere.,
forskellige sekventeringsplatforme
der er en række Illumina-platforme at vælge imellem for at sekvensere cDNA-bibliotekerne.

Billede kredit: Tilpasset fra Illumina
for Forskelle i platformen kan ændre længden af en læser, der genereres, kvaliteten af hits, samt det samlede antal læser sekventeret per løb, og mængden af tid, der kræves for at sekvens bibliotekerne., De forskellige platforme bruger hver en anden Flo .celle, som er en glasoverflade belagt med et arrangement af parrede oligos, der er komplementære til de adaptere, der er tilføjet til dine skabelonmolekyler. Flo .cellen er, hvor sekventeringsreaktionerne finder sted.

Billede kredit: Tilpasset fra Illumina
Sekventering-af-syntese
Illumina sekventering teknologi bruger et sequencer-af-syntese tilgang, som er beskrevet mere detaljeret nedenfor.
i trinnet denatureres DNA-fragmenterne i cDNA-biblioteket og påføres glasstrømscellen., Disse denaturerede fragmenter binder til de komplementære oligoer, der allerede er kovalent bundet til strømningscellebanerne, hvilket resulterer i fastgørelse.
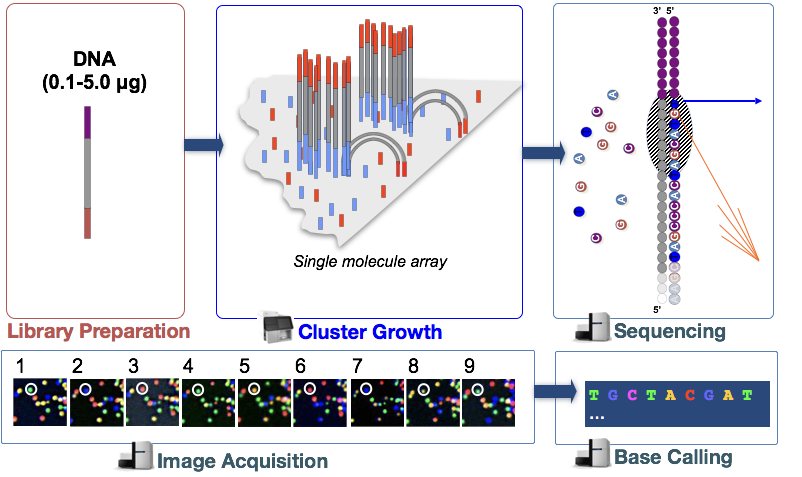
Klyngegenerering
når fragmenterne er fastgjort, begynder en fase kaldet klyngegenerering. Under dette trin forstærkes enkeltfragmenter klonalt for at skabe en klynge (fragmenter i umiddelbar nærhed) af identiske fragmenter. Dette er nødvendigt, således at fluorescensen let kan indfanges fra hver klynge i stedet for et enkelt fragment under nukleotidindarbejdelse i det næste trin.,
- syntetiser komplementet med polymerase
- dsDNA denatureres, og originalt DNA vaskes væk og efterlader syntetiseret streng kovalent bundet til Flo .celle.
- enkeltstrenget hybridiserer med tilstødende adapter til dannelse af en’bro’
- dsDNA udvides med polymerase. Hver streng kovalent bundet til forskellige adapter.
- gentag mange gange for klonalt at forstærke alle unikke fragmenter på Flo .cellen for at danne klynger af identisk sekvens.,
Sekventering af syntese (& image acquisition)
Efter cluster generation, fluorescently-mærkede nukleotider, der er indarbejdet en på et tidspunkt (cyklisk) og fluorescens billeder er taget, til at identificere, hvilke nukleotider bliver indarbejdet i hver klynge i hver cyklus.
- Denatureringsklynger og blok 3′ slutter for at forhindre uønsket priming.Hybridizee sekventering primere til adapter sekvens ved de løse ender.,
- cykle fire NTP ‘ er med fluorescerende markører og terminatorsekvens og polymeraser.
- når NTP er inkorporeret, spændes klyngen af en lyskilde, og der udsendes et karakteristisk fluroscent-signal.
- farven registreres, derefter spaltes terminatoren på farvestof og vaskes. Proces gentages for specifioed antal cyklusser.,
Base Ringer til
Illumina er leverandørejet software, der går gennem alle de billeder, der er taget i den foregående fase og genererer tekst filer med sekvens oplysninger om hver klynge baseret på fluorescens. Ud over at kalde baserne tildeler denne Soft .are en sandsynlighedsscore for at indikere, hvor sikker det handlede om at kalde noget et “A”, Et “t”, et “G” eller et “C”.
Hvis der er uklarheder, f. eks., i en vis cyklus billedet til en klynge, der ikke har en tydelig farve, der kan være forbundet med en bestemt nukleotid, basen ringer software vil have en lav sandsynlighed, der er forbundet med det, og vil tildele en “N” i stedet for “A”, “T”, “G” eller “C”.
I lukning,
- Antallet af klynger ~= Antal læser
- Antallet af sekventering cykler = Længde læser
antallet af cykler (længde læser) vil afhænge af sekventering platform, der anvendes, samt dine præferencer.
NOTE., Hvis du vil udforske sekventering ved syntese mere dybtgående, anbefaler vi denne virkelig dejlige animation tilgængelig på Illuminas YouTube-kanal.
multiple .ing
afhængigt af Illumina-platformen (mise., Hise., ne .tse.) varierer antallet af baner pr. Prøve (dvs. sekvensdybden) og derefter baseret på den platform, du vælger, beregne, hvor mange samlede baner du har brug for til dit sæt prøver., Vi vil tale mere om overvejelser, når vi træffer denne beslutning i den næste lektion om eksperimentelle overvejelser
typisk er gebyrer for sekventering pr. Illumina har derfor udtænkt en dejlig multiplexing metode, der giver mulighed biblioteker fra flere prøver for at samles og planlægges samtidigt på samme bane i en flow-celle. Denne metode kræver tilføjelse af indekser (inden for Illumina-adapteren) eller specielle stregkoder (uden for Illumina-adapteren) som beskrevet i skematikken nedenfor.,
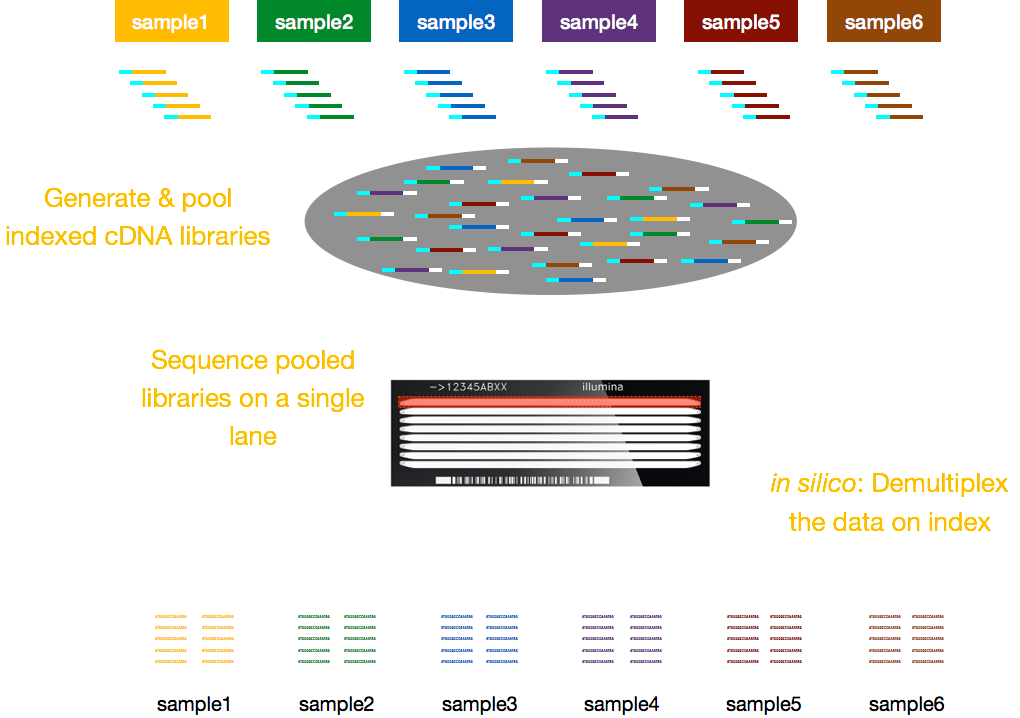
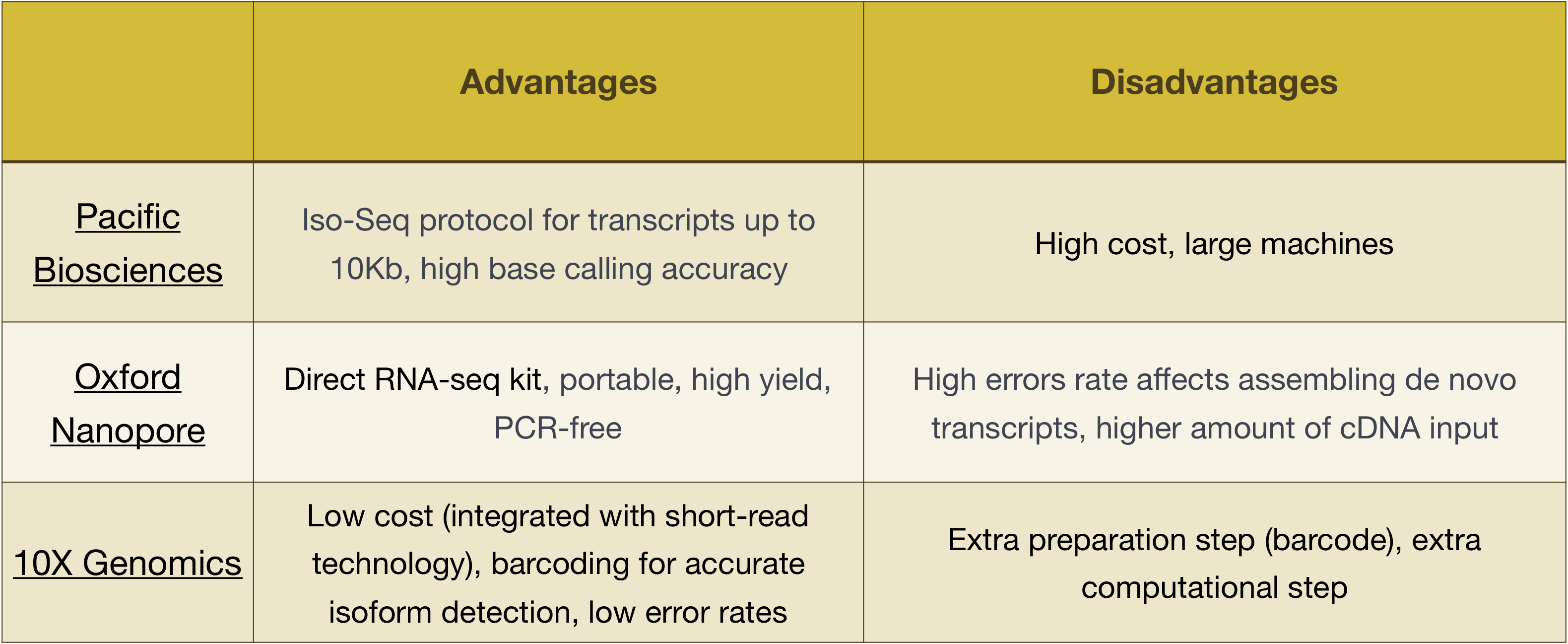
BEMÆRK: workflow, der præsenteres i denne lektion er specifikke for Illumina-sekventering, som i øjeblikket er den mest benyttede sekventering metode., Men der er andre lang-læs sekventering metoder, der er værd at bemærke, sådan som:
- Pacific Biosciences: http://www.pacb.com/
- Oxford Nanopore (MinION): https://nanoporetech.com/
- 10X Genomics: https://www.10xgenomics.com/
Fordele og ulemper ved disse teknologier, der kan udforskes i tabellen nedenfor:
Skriv et svar