
genetisch polymorfisme is het bestaan van alternatieve Staten van DNA, die de variatie van de hogere niveaus van integratie van het organisme bepalen. Er zijn verschillende soorten genoomwijzigingen (mutaties). De meest bestudeerde zijn nucleotide substituties in codage en regelgevende gebieden.
definitie
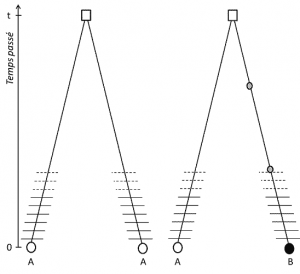
genetisch polymorfisme is het bestaan, in een populatie, van verschillende alternatieve toestanden van DNA, of allelen, op een bepaalde positie in het genoom, of locuspositie van het gen op het chromosoom., In populatiegenetica, een set van homologe genen (homologie klasse). Twee genen zijn homoloog als ze overeenkomen met meiose. Meervoud: loci.. Deze definitie heeft verschillende aspecten:
(1) Ten eerste moet de eigenschap door de chromosomen worden gedragen en overdraagbaar zijn.
(2) dan moeten de allelen homoloog zijn.twee chromosomen of twee genen zouden homoloog zijn als ze overeenkomen en elkaar uitsluiten van meiose. voor hun positie in het genoom, die bepaalde soorten variatie uitsluit die later zullen worden besproken.,
(3) maar omdat de eigenschap overdraagbaar is, houdt positiehomologie ook in dat de allelen homoloog zijn door afstamming; als ze verschillend zijn, is het daarom dat er een mutatie (tenminste) is opgetreden in een van de lijnen die hen verbindt met hun laatste gemeenschappelijke voorouder (figuur 1).
(4) ten slotte kan genetisch polymorfisme worden gedefinieerd op de schaal van de kleinste eenheid waaruit DNA is samengesteld: de nucleotideplaats., Als gevolg hiervan kan elke nucleotidevariant het polymorfisme bepalen van de hogere niveaus van biologische structuur – het gen, eiwit en fenotype van het individu-die schalen worden waarop genetisch polymorfisme kan worden beschreven (Figuur 2).
neem het voorbeeld van het ABO-bloedsysteem, dat overeenkomt met antigene motiesdeel van het antigeen dat wordt herkend door een antilichaam of lymfocytenreceptor. Ook bekend als epitope of antigene determinant. Het zelfde antigeen kan verscheidene epitopes (identiek of verschillend) hebben en zo een gevarieerde immune reactie veroorzaken., betrokken bij de compatibiliteit tijdens bloedtransfusies. Dit is een geval van genetisch polymorfisme huidig in menselijke populaties, waarbij het eiwit ABO glycosyltransferaseenzyme de overdracht van residuen toestaat die suikers aan proteã nen bevatten. In het geval van het ABO-systeem zorgen glycosyltransferase A en B ervoor dat een individu behoort tot groep A of B. Wanneer beide glycosyltransferasen aanwezig zijn, behoort het individu tot groep AB. die drie allelen heeft, A, B en O. een individu kan (AA), (AO); (BB), (BO); (OO) of (AB) genotypen hebben., Zijn fenotype zal dan zijn,,;,; of, waar we zien dat A en B “domineren” O (dat wil zeggen dat hun uitdrukking maskeert die van O), en dat ze onderling co-dominant zijn (het heterozygote tussen A en B heeft een herkenbare uitdrukking,).
een polymorfisme kan eerst worden beschreven op de schaal van de DNA-sequentie van de locus die een eiwit codeert. Sommige polymorfismen zijn” synoniem”, d.w.z. zij veranderen de aminozuurvolgorde van het eiwit niet; zij zijn vaak de talrijke polymorfismen. De anderen wijzigen aminozuren en worden “Vervangende” polymorfismen genoemd., Vervangende polymorfismen van ABO glycosyltransferase zijn van twee types: polymorfismen die het aminozuur veranderen maar niet de antigene eenheid (ze interfereren niet met het ABO fenotype van individuen); en Andere Vervangende polymorfismen, die de ABO fenotypeset van waarneembare kenmerken van een individu bepalen..
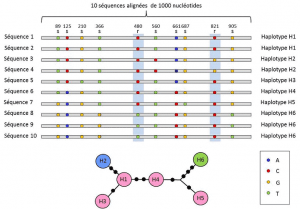
tussen het nucleotide-en het allelniveau wordt bij genetische analyses rekening gehouden met een intermediair beschrijvingsniveau: de lineaire rangschikking van variabele plaatsen op de locus. Deze regelingen, genoemd haplopesgroep van allelen van verschillende loci op hetzelfde chromosoom gevestigd en gewoonlijk samen overgebracht. Haplotype is een vorm van de samentrekking van de Engelse zin haploïde genotype, of haploïde genotype., Alle genen die zich op hetzelfde chromosoom bevinden en waarvan de allelen tijdens de meiose samen scheiden, vormen een haplotype. Deze genen worden “genetisch gekoppeld” genoemd, zijn nuttig in de evolutionaire genetica omdat ze het mogelijk maken om de genealogische verbanden tussen de allelen te vinden (Figuur 2).
Nucleotidepolymorfismen bestaan ook in intergene gebieden (buiten coderingsgebieden). Sommigen beà nvloeden de gebieden waar de genuitdrukking wordt geregeld en daarom phenotypic uitdrukking hebben. Anderen hebben geen bekend effect en worden aangeduid als “stil”.,
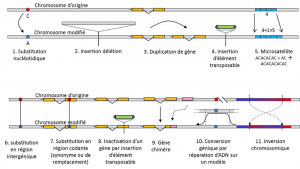
naast nucleotidesubstituties verwijst polymorfisme voor een bepaald type mutatie, microsatellieten, naar variaties in de lengte van een vaak niet-coderend DNA-fragment (Figuur 3) als gevolg van een verandering in het aantal herhalingen in een korte herhaalde sequentie van nucleotiden, bijvoorbeeld CACACACA of TGTGTG.
niet alle genetische variatie valt onder de definitie van genetisch polymorfisme, aangezien hiervoor de positiehomologie van alternatieven vereist is (Figuur 3)., Aldus, zijn er herhaalde opeenvolgingen verkregen door het dupliceren van een gen, waarvoor twee herhalingen niet homologe per positie zijn. Vaak kunnen we ook niet praten over homologie voor transposeerbare elementen, omdat ze over het algemeen van positie veranderen tijdens celgeneraties, zich kunnen vermenigvuldigen en het genoom kunnen binnendringen. De homologie van twee transposeerbare elementen kan dan niet worden gedefinieerd. Aan de andere kant komen twee codeersequenties van dezelfde locus, waarvan er één intact is, en de andere geïnactiveerd door het inbrengen van een transposeerbaar element, goed overeen met een homologiesituatie., Op een dag zullen we waarschijnlijk overwegen om te praten over polymorfisme voor de modificaties van zogenaamde “epigenetische” chromosomen, die soms worden overgedragen over meerdere celgeneraties, somatickwalificeert niet-reproductieve cellen, of soma. Een somatische mutatie, die een gen van een somatische cel beïnvloedt, verdwijnt met de drager individu. of kiem kwalificeert gameten. Een kiemlijn mutatie kan worden overgedragen aan zijn nakomelingen., zoals methylering. Hun belang in de evolutie van populaties moet nog worden beoordeeld., De beperking van de notie van genetisch polymorfisme tot bepaalde categorieën van variatie – in wezen nucleotide substituties – komt uit het grote nut van de laatste in het verkennen van de geschiedenis van natuurlijke populaties. Ze lenen zich voor wiskundige modellering van evolutie.
het woord polymorfisme betekent “verschillende vormen”. Het is tegengesteld aan monomorfisme, wat de afwezigheid van variatie aangeeft. In de woordenschat van de biologie wordt deze tegenstelling al lang, vóór de genetica, gebruikt om de coëxistentie van verschillende soorten individuen in dezelfde soort aan te geven, bijv., kaste polymorfisme (koningin en arbeiders) bij sociale insecten; seizoensgebonden polymorfisme (vachtveranderingen) bij sommige Arctische zoogdieren. Deze gevallen vallen niet onder genetisch polymorfisme, een recenter concept met een engere en preciezere betekenis. Continue variaties (bijvoorbeeld variatie in grootte) vallen ook niet onder de categorie polymorfisme, omdat ze geen verschillende alternatieven bieden. Locus die de grootte beïnvloeden, vallen echter binnen deze definitie., We zullen hieronder de meest bestudeerde genetische variaties bespreken omdat ze het belangrijkst zijn voor evolutie, nucleotide substituties, en dan zullen we de variatie van fenotypen bespreken.
meting
Het volstaat niet te zeggen dat een locus min of meer variabel is zonder een kwantitatieve schatting van dit arrest te geven. Een onderzoeker kan de variatie op verschillende schalen bestuderen. Als hij alleen geïnteresseerd is in de allelen van een eiwit, zal hij de allelische diversiteit meten, gesymboliseerd door ‘H’. Als hij geïnteresseerd is in DNA diversiteit, zal hij nucleotide diversiteit meten, gesymboliseerd door ‘π’.,
De alleldiversiteit H wordt gedefinieerd als de kans dat twee verschillende allelen worden getrokken tijdens twee sampling met vervangingperformeren van een opeenvolgende loting met levering van P tokens in een urn die n tokens bevat, betekent het nemen van een eerste token, het lezen van de waarde, het terugzetten in de urn, het nemen van een tweede token, het lezen van de waarde, het terugzetten in de urn, enz. tot de PTH token. Dit betekent het kiezen van P objecten tussen n met herhaling (u kunt hetzelfde object meerdere keren kiezen) en in volgorde (de volgorde waarin u de objecten kiest is belangrijk)., Het aantal opeenvolgende trekkingen met tokens onder n is: n × n × n × … × n = np.. Als we pi de frequentie van het allel van rang i noemen, tonen we aan dat de kans om twee keer hetzelfde allel te kiezen F = Σ pi2 is. De allelische diversiteit van het monster is dan de aanvulling op 1, d.w.z.:
H = 1 – Σ pi2 (1)
Dit geldt voor zowel eiwitallelen als haplotypes. Dit heet haplopieke diversiteit. Het kan ook heterozygositeit worden genoemd, omdat in het geval van een diploïde locus, het de verwachte frequentie van heterozygoten geeft.,
De nucleotidediversiteit π is het equivalent van H op de schaal van elk nucleotide. Het wordt berekend als het gemiddelde van het aantal nucleotideverschillen tussen opeenvolgingen in een steekproef die twee door twee wordt genomen (δij), gedeeld door de lengte van het fragment van DNA in het aantal nucleotiden (L).
π = gemiddelde (δij)/L (2) (zie referentie )
deze waarde verschilt per soort. In de codagedelen van het menselijke genoom, verschillen twee willekeurig gekozen chromosomen uit de bevolking gemiddeld door één nucleotide per duizend., Bij de fruitvlieg (Drosophila melanogaster) is dit verschil ongeveer een op de honderd. De vlieg is dus tien keer meer variabel dan de mens. Wanneer deze waarden gerelateerd zijn aan de grootte van de codeergebieden (ongeveer 15.500 genen in Drosophila, minstens 22.000 in de mens), of zelfs genomen (140 miljoen basenparen per haploïde genoom in vliegen, ongeveer 3.200 miljoen of twintig keer meer in de mens), is het aantal polymorfe sites astronomisch en resulteert in niemand die geboren wordt uit de geseksualiseerde generatie in een soort is, is en zal genetisch identiek zijn aan een ander verleden, heden of toekomst., De informatiekracht van DNA-polymorfisme is immens. In forensisch onderzoek kunnen detectives elke verdachte identificeren van zestien microsatelliet locussen.
geschiedenis
De term variatie komt voor in de titel van de eerste twee hoofdstukken van Darwin ‘ s boek The Origin of Species (1859). Darwin introduceerde dit begrip in de natuurwetenschappen op een moment dat de wetten van biologische erfelijkheid een mysterie bleven, en wijdde er een ander belangrijk boek aan, Variation in Animals and Plants (1868)., Ervan overtuigd dat evolutie ging over kleine variaties die weinig verschil maakten voor de aanpassing van organismen aan hun leefomstandigheden, hechtte hij cruciaal belang aan kleine kwantitatieve variaties, wat zijn opvolgers (met name Karl Pearson) ertoe bracht de biometryScience van levensmeting te vinden. Verwijst in zeer brede zin naar de kwantitatieve studie van levende wezens.. Maar de herontdekking van Mendels wetten in 1900 veranderde de interesse naar discontinue variaties.
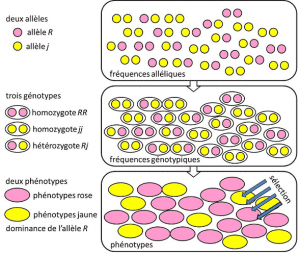
van 1908 tot 1930 was populatiegenetica in wezen een theoretische discipline die Darwinisme met Mendeliaanse erfelijkheid wilde verzoenen. Verwijst naar een genetische eigenschap met eenvoudig determinisme, door een paar of een klein aantal paren van genen. en waar kansen een grote rol speelden. Genetica is contra-intuïtief., Het voorspelt dat er geen reproductie van de ouder in zijn afstammeling is, omdat het genotype van de laatste het resultaat is van de loterij van de scheiding van de allelen vóór de fusie van de twee ouderlijke half-genomen. Men realiseerde zich dat dit de allelische frequentie is waarbij het allel van een variant wordt gevonden in een populatie. Uitgedrukt als percentage of percentage. De som van de allelfrequenties van alle allelen van een gen in een populatie is daarom per definitie gelijk aan 1. In de populatiegenetica vertegenwoordigen allelfrequenties de genetische diversiteit op het niveau van de populatie of de soort., die tussen generaties worden overgedragen, niet genotypen of fenotypen. Deze frequenties zijn min of meer stabiel van generatie op generatie en produceren, voorbij de meervoudige verstrengeling van afstammelingen tussen individuen, dezelfde genotypische frequenciesgenetische structuur van de populatie. Bepaald aan de hand van de allelische frequenties (Figuur 4). De verdeling van populatiegenotypen, geassocieerd met variantie, is daarom de enige voorspelbare factor., Rond 1930 hielpen de drie theoretici Ronald Fisher, JBS Haldane en Sewall Wright om het begrip van de Mendelse bevolkingspopulatie te creëren waarvan de erfelijkheid Mendels wetten gehoorzaamde. . In deze context associeert evolutie noodzakelijkerwijs drie structurele niveaus: het gen, het individu en de populatie. Deze Drieweg associatie kan worden samengevat met de volgende formule: populatieevolutie is een verandering in allelfrequentie (genschaal, maar ook groepsschaal), een verandering die afhangt van het sorteren van fenotypen door selectie (individuele schaal)., Empirisch populatiegenetisch onderzoek werd toen ontwikkeld, maar werd lange tijd beperkt door onze onwetendheid over hoe chromosomen werken (de structuur van DNA werd begrepen in 1953 en de sequencing ervan begon in 1977), en moest terugvallen op de studie van de weinige zichtbare polymorfismen, zoals de kleuring van lieveheersbeestjes dekschilden of de schelp van slakken (zie Focus The great slak debate). Er was veel discussie onder genetici over de vraag of natuurlijke populaties over het algemeen polymorf of monomorf waren, en of polymorfisme op zich wel of niet gunstig was., De studie van macromoleculaire schaalvariatie moest wachten tot 1966 (voor eiwitten) en 1983 (voor DNA) alvorens deze vragen te beantwoorden.
kwantitatieve variaties en erfelijkheid
wanneer we om ons heen kijken, zien we fysieke verschillen tussen wezens, sommige complexe gelaatstrekken, andere gemakkelijk te meten zoals gewicht of lengte. Het gezond verstand suggereert dat ze gedeeltelijk overdraagbaar zijn, zij het op een manier die moeilijk te specificeren is. Deze eigenschappen zijn vaak polygeen onder de afhankelijkheid van vele genen. We hebben het over polygene erfelijkheid. Diabetes is een polygene ziekte.,, wat betekent dat ze worden beïnvloed door vele locussen. Ontwikkelingsgenetica, en de toepassing ervan op de vergelijking van soorten (evo-devo, voor “evolutie van ontwikkeling”) ontrafelen het web van complexe interacties die ervoor zorgen dat tijdens de ontwikkeling de cellen van het lichaam, allemaal met identieke genetische bagage, zich onderscheiden door epigenese om verschillende weefsels te geven. Het is nog onduidelijk hoe complexe karakters worden gebouwd. Meetbare fenotypen kunnen worden bestudeerd met behulp van biometrie, gebaseerd op de eigenschappen van statistische distributies., Een waarde met nuttige eigenschappen is de variantiein een steekproef van individuen waarin een bepaalde eigenschap werd gemeten, is de variantie het verschil tussen het gemiddelde van het kwadraat van de waarden en het kwadraat van het gemiddelde van de waarden. Deze maatregel, die altijd positief is, wijst op de verspreiding van individuen. . Wanneer verschillende onafhankelijke oorzaken de variatie van een lijn bepalen, zijn hun varianties additief en hun som geeft de variantie van de lijn. Als ze niet onafhankelijk zijn, wordt de som van de covarianties opgeteld bij de som van de varianties., De totale variantie Vt van een fenotypische eigenschap is zodanig dat :
Vt = Vga+Vgd+Vgd+VGI+Ve
Vga, additieve genetische variantie, de som is van de varianties die aan elke locus worden veroorzaakt, onafhankelijk van de andere; VGD, dominante genetische variantie, resultaten van de interactie van de twee allelen van dezelfde locus; VGI, interactievariantie tussen locussen, resultaten uit het feit dat de Locussen van hetzelfde individu interageren; ve, variantie van de omgeving, wordt hier verondersteld onafhankelijk van genetische variantie.,
de genetische variantie van dominantie is gemeenschappelijk voor twee kinderen van dezelfde ouders, omdat ze dominantie-effecten delen wanneer ze op een bepaalde locus dezelfde allelen van beide ouders hebben gekregen, wat bij hen gebeurt bij één op de vier locus. Ze delen deze effecten niet met hun ouders, dus ze lijken meer op elkaar dan op hun ouders, ondanks het feit dat ze zoveel genen met elkaar delen. Zeker, een ouder kan, bij toeval, dezelfde twee allelen hebben gekregen als een van zijn kinderen op een bepaalde locatie., Deze kans hangt af van de frequentie van allelen in de populatie. We zien dat de mate van gelijkenis tussen een kind en zijn of haar ouder voor een bepaalde eigenschap een openbaar bezit is.
meer in het algemeen kunnen alle componenten van genetische variantie (Vga+Vgd+Vgi) per populatie verschillen. In foklijnen is inteelt een veel voorkomend geval, wat de genetische variantie uitputt: als gevolg daarvan kan een eigenschap hetzelfde gemiddelde hebben in twee populaties, maar een andere genetische variatie hebben. Het vermogen van een kweker om een eigenschap te selecteren is daarom een kenmerk van de lijn, niet van de eigenschap.,
de relatie tussen ouders en kinderen wordt gemeten aan de hand van erfelijkheid h2. Het wordt gedefinieerd als de verhouding tussen additieve genetische variantie en totale variantie :
h2 = Vga/Vt

(2) correlatie van 0, waarbij de grootte van de kinderen onafhankelijk is van die van de ouders. Een waardevolle gemiddelde ouder heeft een kind van onverschillige waarde voortgebracht, binnen de verdeling van kinderen. De coördinaten van de punten bevinden zich op een cirkel; de verwachting van hun waarde bevindt zich op een lijn evenwijdig aan de as van de ouders (d)., (3) tussenliggende ouder-kind correlatie tussen 0 en 1: de coördinaten van de punten bevinden zich op een ellips. De verwachting van hun waarde bevindt zich niet langer op de doorsnede van de coördinaatassen (die ook de hoofdas van de distributie-ellips is), maar op de regressielijn r. een gemiddelde ouder van waarde heeft een kind van waarde b (tussen 0 en a) met een bepaalde variantie voortgebracht. Opgemerkt wordt dat geval (3) tussen de gevallen (1) en (2)
Het is weergegeven onder verschillende veronderstellingen in Figuur 5., Erfelijkheid is het vermogen om te bepalen of een functie kan worden geselecteerd. Figuur 5-3 laat zien dat een kweker in staat zal zijn, binnen een kruising generatie, om het gemiddelde van een karakter te verplaatsen van de waarde 0 naar de waarde b als hij individuen selecteert met waarde a. We kunnen aantonen dat b = h2a. om een eigenschap te selecteren, is het nuttig om de erfelijkheidsratio te verhogen door het milieu te standaardiseren (waardoor de variatie in de omgeving wordt verminderd), en door zich te plaatsen in de omgevingsomstandigheden die het uiterlijk van de eigenschap bevorderen., De geneticus Waddington heeft aangetoond dat een verandering van omgeving in de natuur nieuwe eigenschappen kan onthullen die vervolgens tijdens de evolutie zullen worden geselecteerd.
referenties en opmerkingen
omslagafbeelding. Slakkenhuis Cepaea nemoralis & Cepaea hortensis. André Künzelmann, UFZ.,
Deze formule kan op een meer conventionele manier worden geschreven als: \pi =\frac{n(n-1)} {2L}\sum _{i=1}^{n-1}\sum_{j=i+1}^{n}\delta ij
omdat het een steekproef is van individuen bij wie de bestudeerde eigenschap werd gemeten, kan variantie worden gedefinieerd als het verschil tussen het gemiddelde van het kwadraat van de waarden en het kwadraat van het gemiddelde van de waarden. Deze maatregel, die altijd positief is, wijst op de verspreiding van individuen.
Dit is erfelijkheid in de strikte zin van het woord. Erfelijkheid in brede zin is de verhouding tussen de som van de genetische varianties en de totale variantie., Deze formule geeft erfelijkheid met beide ouders (de “gemiddelde ouder”); de schatting met slechts één ouder zou H2/2 geven.
The Environmental Encyclopedia of the Environment by the Association des Encyclopedia de l ‘environnement et de l’ énergie (www.a3e.fr), contractueel verbonden met de Universiteit van Grenoble Alpes en Grenoble INP, en gesponsord door de Franse Academie van Wetenschappen.
om dit artikel te citeren: VEUILLE Michel (2021), Genetic polymorphism and variation, Encyclopedia of the Environment, url : https://www.encyclopedie-environnement.org/en/life/genetic-polymorphism-and-variation/.,
de artikelen in de Encyclopedia of the Environment worden beschikbaar gesteld onder de voorwaarden van de Creative Commons BY-NC-SA licentie, die reproductie toestaat onder voorbehoud van: het citeren van de bron, het niet commercieel gebruik ervan, het delen van identieke initiële voorwaarden, het reproduceren bij elke hergebruik of distributie van de vermelding van deze Creative Commons BY-NC-SA licentie.
Geef een reactie