durata Aproximativă: 90 de minute
Obiective de Învățare:
- Descrie procesul de ARN-seq biblioteca de pregătire
- Descrie Illumina metoda de secvențiere
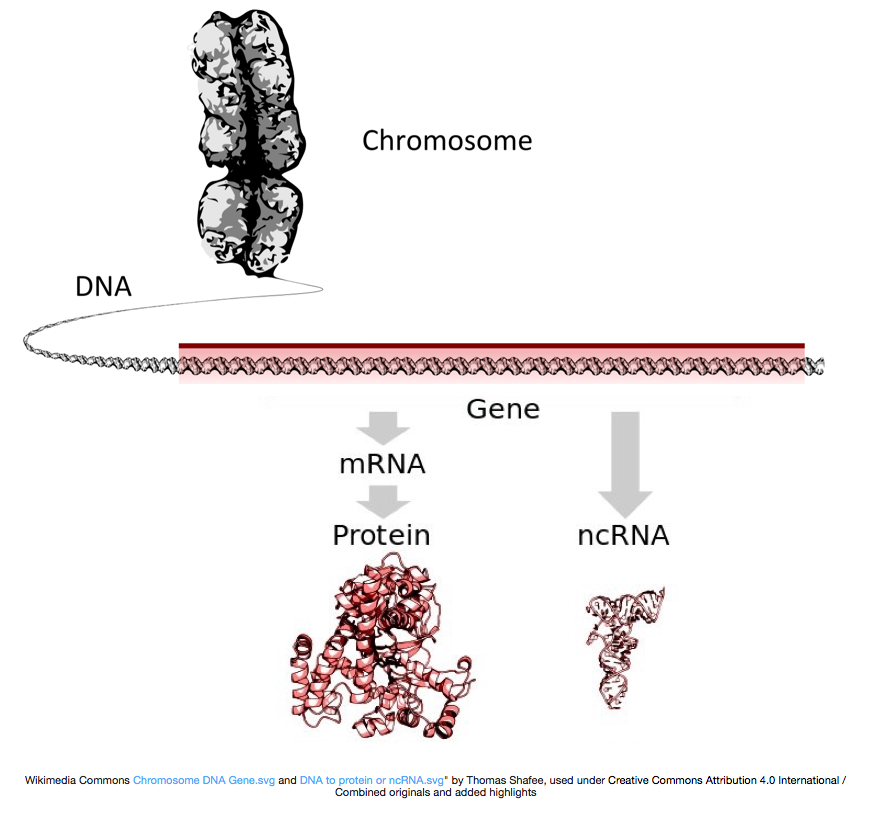
Introducere a ARN-urm
ARN-seq este un joc interesant de tehnică experimentală, care este utilizată pentru a explora și/sau cuantificarea expresiei genice în cadrul sau între condiții.,după cum știm, genele oferă instrucțiuni pentru a face proteine, care îndeplinesc o anumită funcție în interiorul celulei. Deși toate celulele conțin aceeași secvență ADN, celulele musculare sunt diferite de celulele nervoase și de alte tipuri de celule din cauza diferitelor gene care sunt activate în aceste celule și a diferitelor ARN-uri și proteine produse.diferite procese biologice, precum și mutații, pot afecta care gene sunt activate și care sunt dezactivate, în plus față de cât de multe gene specifice sunt activate/dezactivate.,pentru a produce proteine, ADN-ul este transcris în ARN mesager sau ARNm, care este tradus de ribozom în proteină. Cu toate acestea, unele gene codifică ARN care nu se traduce în proteine; aceste ARN-uri sunt numite ARN-uri care nu codifică sau ncRNAs. Adesea, aceste ARN-uri au o funcție în sine și includ ARN-uri, tRNAs și siRNAs, printre altele. Toate ARN-urile transcrise din gene se numesc transcrieri.
pentru a fi tradus în proteine, ARN-ul trebuie să fie supus procesării pentru a genera ARNm., În figura de mai jos, partea superioară a imaginii reprezintă o genă din ADN, formată din regiunile netraduse (UTRs) și Cadrul deschis de citire. Genele sunt transcrise în pre-ARNm, care conține încă secvențele intronice. După prelucrarea post-transcipțională, intronii sunt îmbinați și se adaugă o coadă polyA și un capac de 5′ pentru a obține transcrieri ARNm mature, care pot fi traduse în proteine.,
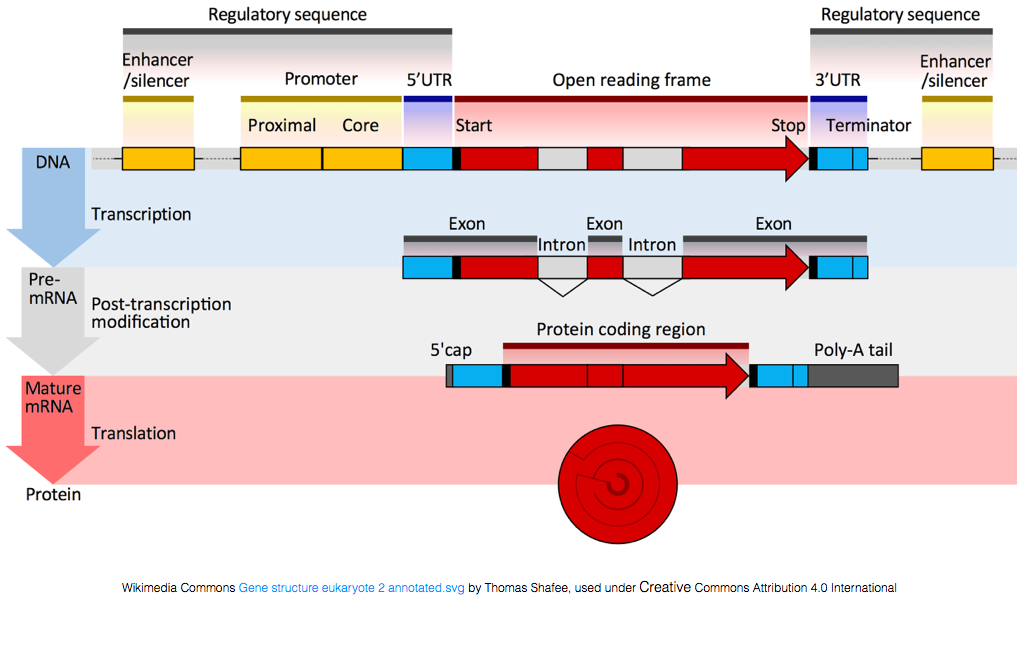
în timp ce transcrierile ARNm au o coadă polyA, multe dintre transcrierile ARN care nu codifică nu, deoarece procesarea post-transcripțională este diferită pentru aceste transcrieri.
Transcriptomică
transcriptomul este definit ca o colecție a tuturor citirilor de transcriere prezente într-o celulă., ARN-următoarele date pot fi folosite pentru a explora și/sau cuantificarea transcriptomului a unui organism, care pot fi utilizate pentru următoarele tipuri de experimente:
- Diferențial Gene Exprimare: cantitative de evaluare și comparare al transcriptului
- Transcriptomului de asamblare: construirea profilului de transcris regiuni ale genomului, o evaluare calitativă.,
- Poate fi folosit pentru a ajuta la construirea mai bine gene modele, și de a le verifica, folosind adunarea
- Metatranscriptomics sau comunitate transcriptomului analiza
Illuminati biblioteca de pregătire
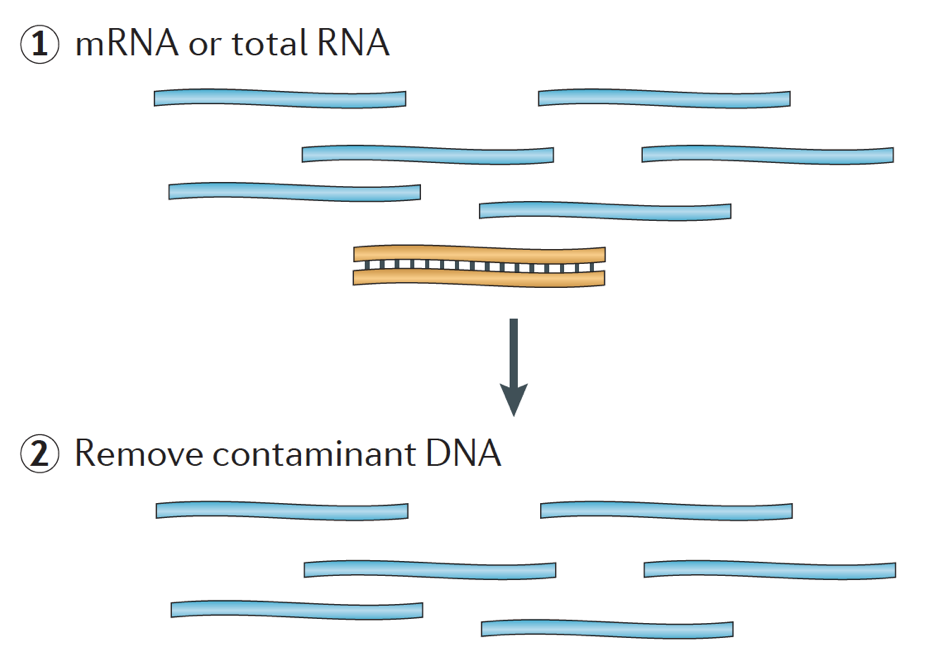
atunci Când începe un ARN-seq experiment, pentru fiecare probă ARN-ul trebuie să fie izolate și transformat într-un adnc biblioteca pentru secvențiere. Fluxul general de lucru pentru pregătirea bibliotecii este detaliat în imaginile pas cu pas de mai jos.
pe scurt, ARN-ul este izolat din probă și ADN-ul contaminant este îndepărtat cu Dnază.,
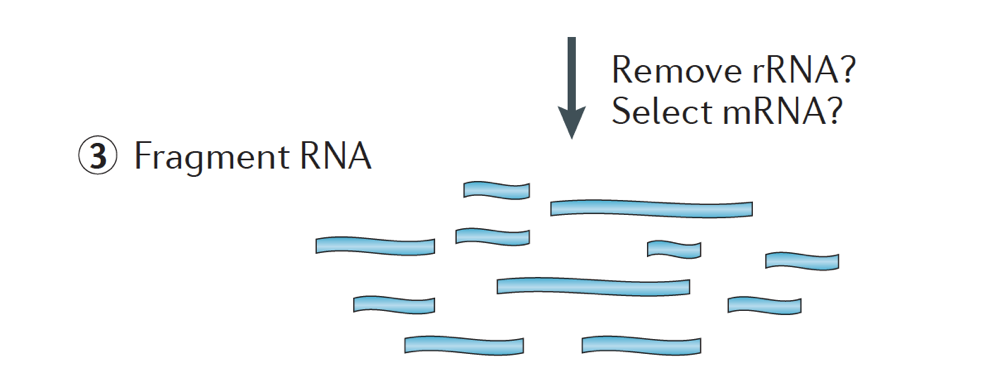
proba de ARN este apoi supusă fie selecției ARNm (selecția polyA), fie epuizării ARNr. ARN-ul rezultat este fragmentat.
în General, ARN ribosomal reprezintă majoritatea RNAs prezent într-o celulă, în timp ce mesagerul RNAs reprezintă un procent mic din totalul ARN-ului, ~2% din oameni. Prin urmare, dacă vrem să studiem genele care codifică proteinele, trebuie să îmbogățim pentru ARNm sau să epuizăm ARNr., Pentru analiza expresiei genelor diferențiale, cel mai bine este să îmbogățiți pentru Poli(a)+, cu excepția cazului în care intenționați să obțineți informații despre ARN-uri lungi care nu codifică, apoi faceți o epuizare a ARN ribozomal.
dimensiunea țintă fragmente în finală biblioteca este un parametru cheie pentru construcția bibliotecă. Fragmentarea ADN-ului se face de obicei prin metode fizice (de exemplu, acustice forfecare și sonicare) sau metode enzimatice (de exemplu, non-specifice endonucleaze cocktail-uri și transposase tagmentation reacții.,
ARN-ul este inversa apoi transcrise în dublu catenar adnc și secvența de adaptoare sunt apoi adăugate la capetele fragmentelor.
bibliotecile ADNc pot fi generate într-un mod de a reține informații despre ce catenă de ADN a fost transcris ARN-ul. Bibliotecile care păstrează aceste informații se numesc biblioteci eșuate, care sunt acum standard cu Truseq truseq stranded RNA-Seq kituri Illumina., Bibliotecile blocate nu ar trebui să fie mai scumpe decât cele neîngrădite, deci nu există niciun motiv pentru a nu obține aceste informații suplimentare.,
Exista 3 tipuri de izoenzime biblioteci disponibile:
- Înainte (secondstrand) – citește seamănă cu secvența de gene sau secondstrand adnc secvența
- Inversă (firststrand) – citește seamănă cu complement de secvența de gene sau firststrand adnc secvență (TruSeq)
- Unstranded
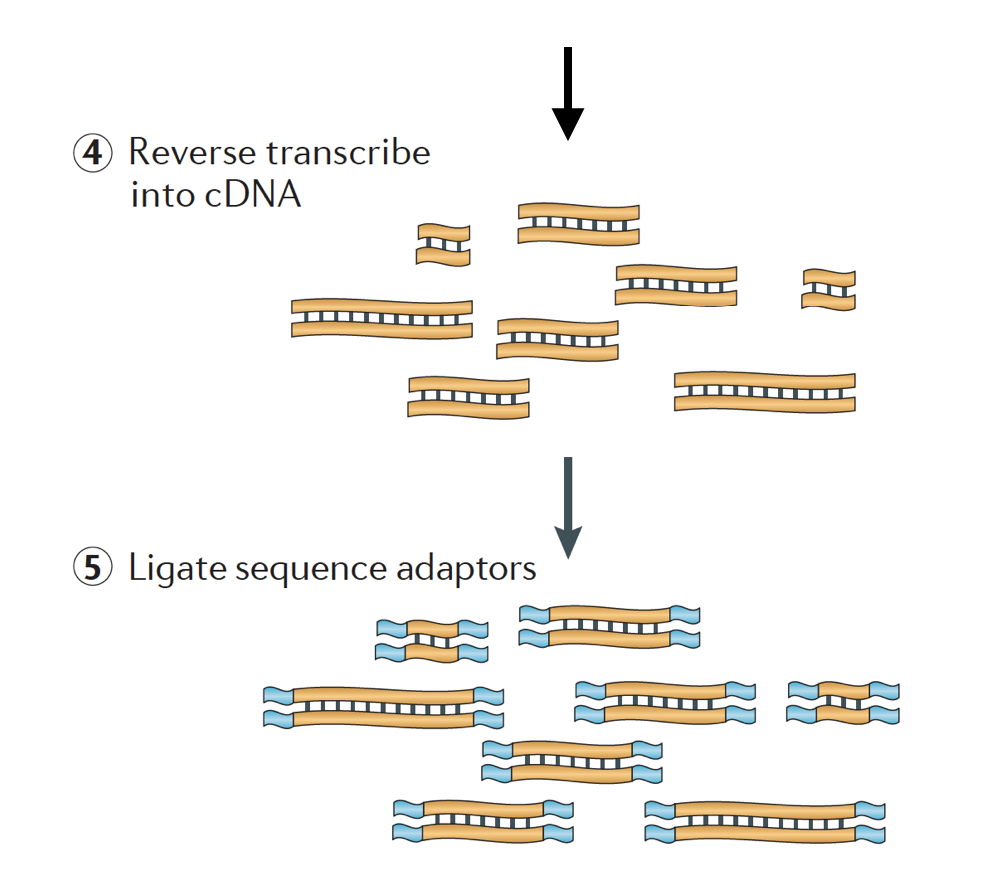
în cele din Urmă, fragmentele sunt PCR amplificat, dacă este necesar, și fragmentele sunt dimensiunea selectată (de obicei ~300-500bp) pentru a termina bibliotecă.
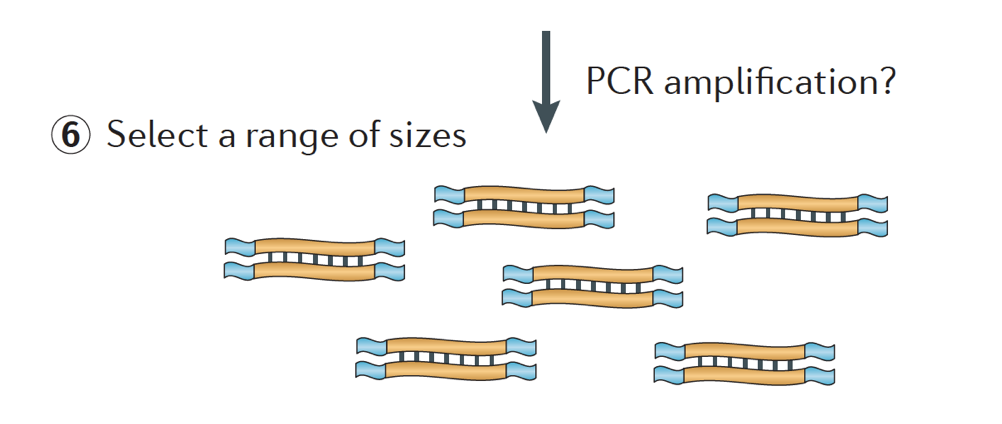
Imaginea de credit: Martin J. A. și Z. Wang, Nat. Rev., Genet. (2011) 12:671-682
Illumina Sequencing
Single-end versus Paired-end
după pregătirea bibliotecilor, secvențierea poate fi efectuată pentru a genera secvențele nucleotidice ale capetelor fragmentelor, care sunt numite citește. Veți avea posibilitatea de a alege secvențierea unui singur capăt al fragmentelor ADNc (citire cu un singur capăt) sau ambele capete ale fragmentelor (citire cu capăt pereche).,
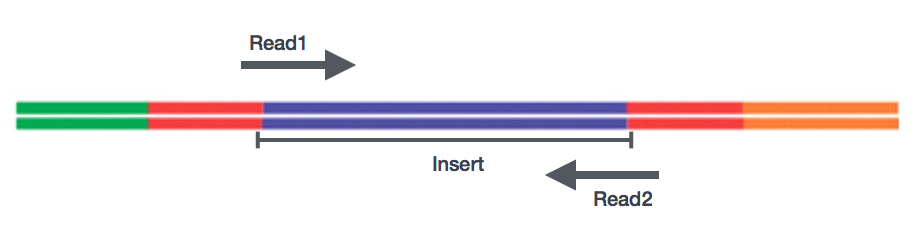
- SE – Singur capăt date => Doar WordPressdb1
- PE – Asociat-end de date => WordPressdb1 + Read2
- poate fi de 2 separate FastQ fișiere sau doar unul cu intercalat perechi
în General, cu un singur scop, secvențiere este suficientă dacă nu este de așteptat ca citeste vor potrivi cu mai multe locații de pe genom (de exemplu, organisme cu multe paralogous gene), ansambluri sunt efectuate, sau pentru îmbinare izoenzimei diferențiere. Fiți conștienți de faptul că citirile de capăt pereche sunt, în general, de 2 ori mai scumpe.,
diferite platforme de secvențiere
există o varietate de platforme Illumina din care puteți alege pentru a secvența bibliotecile cDNA.

Imaginea de credit: Adaptat de la Illuminati
Diferențe în platforma poate modifica lungimea citește generate, în calitate de citește, precum și numărul total de citește esalonate pe fugă și cantitatea de timp necesară pentru secvența de biblioteci., Diferitele platforme utilizează fiecare o celulă de flux diferită, care este o suprafață de sticlă acoperită cu un aranjament de oligos pereche care sunt complementare adaptoarelor adăugate moleculelor șablonului. Celula de flux este locul în care au loc reacțiile de secvențiere.

credit Imagine: adaptat din Illumina
secvențiere prin sinteză
Tehnologia de secvențiere Illumina utilizează o abordare secvențiere prin sinteză care este descrisă mai detaliat mai jos.
în etapa, fragmentele de ADN din biblioteca ADNc sunt denaturate și aplicate celulei fluxului de sticlă., Aceste fragmente denaturate se leagă de oligozii complementari care sunt deja legați covalent de benzile celulare de flux, rezultând atașarea.
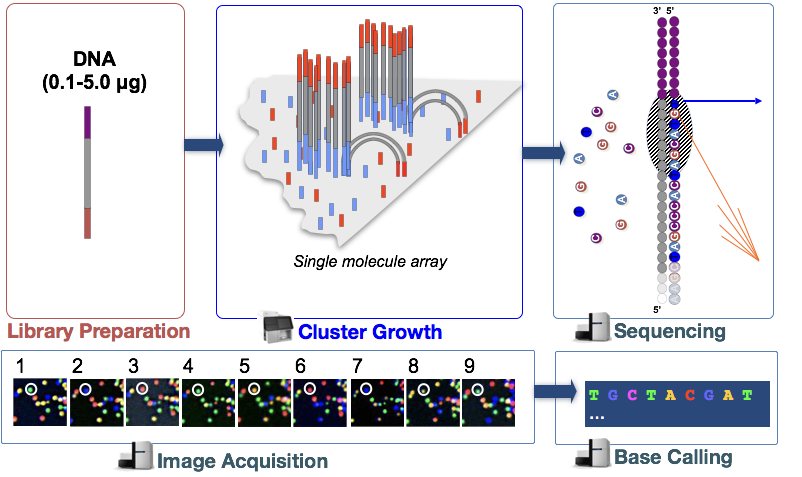
Cluster Generation
odată ce fragmentele s-au atașat, începe o fază numită cluster generation. În timpul acestei etape, fragmente unice sunt amplificate clonal pentru a crea un cluster (fragmente în imediata apropiere) de fragmente identice. Acest lucru este necesar pentru ca fluorescența să poată fi captată cu ușurință din fiecare cluster, în locul unui singur fragment, în timpul încorporării nucleotidelor în etapa următoare.,
- sintetiza complementul cu polimeraza
- dsDNA este denaturat, și ADN-ul original spălat lăsând Strand sintetizat covalent legat de fluxul de celule.
- hibridizează un singur fir cu adaptor adiacent pentru a forma o „punte”
- dsDNA este extins prin polimerază. Fiecare fir legat covalent la diferite adaptor.
- repetați de mai multe ori pentru a amplifica clonal toate fragmentele unice pe celula de flux pentru a forma grupuri de secvență identică.,
sinteza de Secvențiere (& image acquisition)
După cluster generație, fluorescent-a etichetat nucleotide sunt încorporate unul la un moment dat (ciclic) și fluorescență imagini sunt capturate pentru a identifica care nucleotide devine încorporată în fiecare cluster în fiecare ciclu.
- clustere de DENATURĂ și blocul 3′ se termină pentru a preveni amorsarea nedorită.
- hibridizați grundurile de secvențiere la secvența adaptorului la capetele libere.,
- ciclu patru NTPs cu markeri fluorescenți și secvența de terminator și polimeraze.
- odată ce NTP este încorporat, clusterul este excitat de o sursă de lumină și un semnal caracteristic fluroscent este emis.
- culoarea este înregistrată, apoi terminatorul de pe colorant este scindat și spălat. Procesul se repetă pentru numărul specific de cicluri.,
Baza de Asteptare
Illumina are software-ul proprietar care trece prin toate imaginile capturate în etapa anterioară și generează fișiere text cu secvența de informații despre fiecare cluster bazat pe fluorescență. În plus față de apelarea bazelor, acest software atribuie un scor probablity pentru a indica cât de sigur a fost despre apelarea ceva un „A”, Un „T”, un „G” sau un „C”.
dacă există ambiguități, de ex., la un anumit ciclu imaginea pentru un cluster nu are o culoare distinctă care poate fi asociată cu o nucleotidă specifică, software-ul de apelare de bază va avea o probabilitate scăzută asociată cu aceasta și ar atribui un „N” în loc de „A”, „T”, „G” sau „C”.
În încheiere,
- Numărul de clustere ~= Număr de citește
- Numarul de cicluri de secvențiere = Lungimea de citește
numărul de cicluri (lungimea citește) va depinde de secvențiere platforma utilizată precum și preferințele dumneavoastră.
notă., Dacă doriți să explorați secvențierea prin sinteză în profunzime, vă recomandăm această animație foarte drăguță disponibilă pe canalul YouTube Illumina.
Multiplexare
în Funcție de Illuminati platforma (MiSeq, HiSeq, NextSeq), numărul de benzi pe fluxul de celule, și numărul de citește care pot fi obținute de pe banda de circulație variază pe scară largă. Va trebui să decideți câte citiri doriți pe eșantion (adică adâncimea de secvențiere) și apoi, pe baza platformei pe care o alegeți, să calculați câte benzi totale veți avea nevoie pentru setul dvs. de eșantioane., Vom vorbi mai multe despre considerente atunci când luăm această decizie în următoarea lecție despre considerente experimentale
De obicei, taxele pentru secvențiere sunt pe banda celulei de flux și veți putea rula mai multe probe pe bandă. Prin urmare, Illumina a conceput o metodă de multiplexare plăcută, care permite bibliotecilor din mai multe eșantioane să fie reunite și secvențiate simultan în aceeași bandă a unei celule de flux. Această metodă necesită adăugarea de indici (în cadrul adaptorului Illumina) sau coduri de bare speciale (în afara adaptorului Illumina) așa cum este descris în schema de mai jos.,
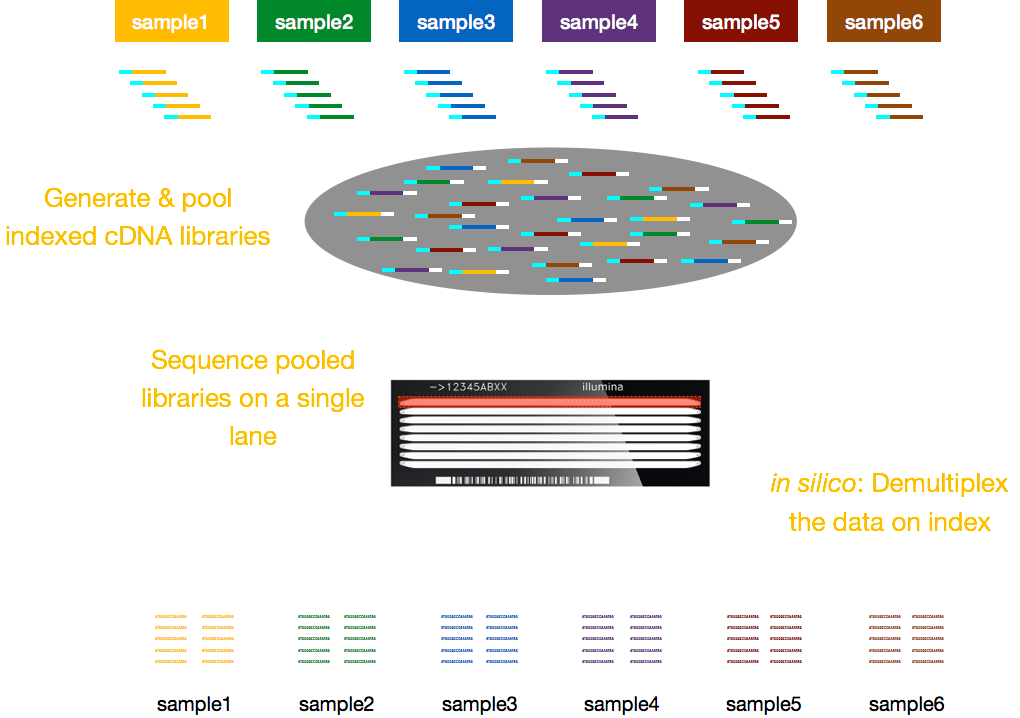
NOTĂ: fluxul De lucru prezentate în această lecție este specific Illuminati secvențiere, care este în prezent cea mai utilizată metoda de secvențiere., Dar există și alte lungă citit secvențiere metode de remarcat, cum ar fi:
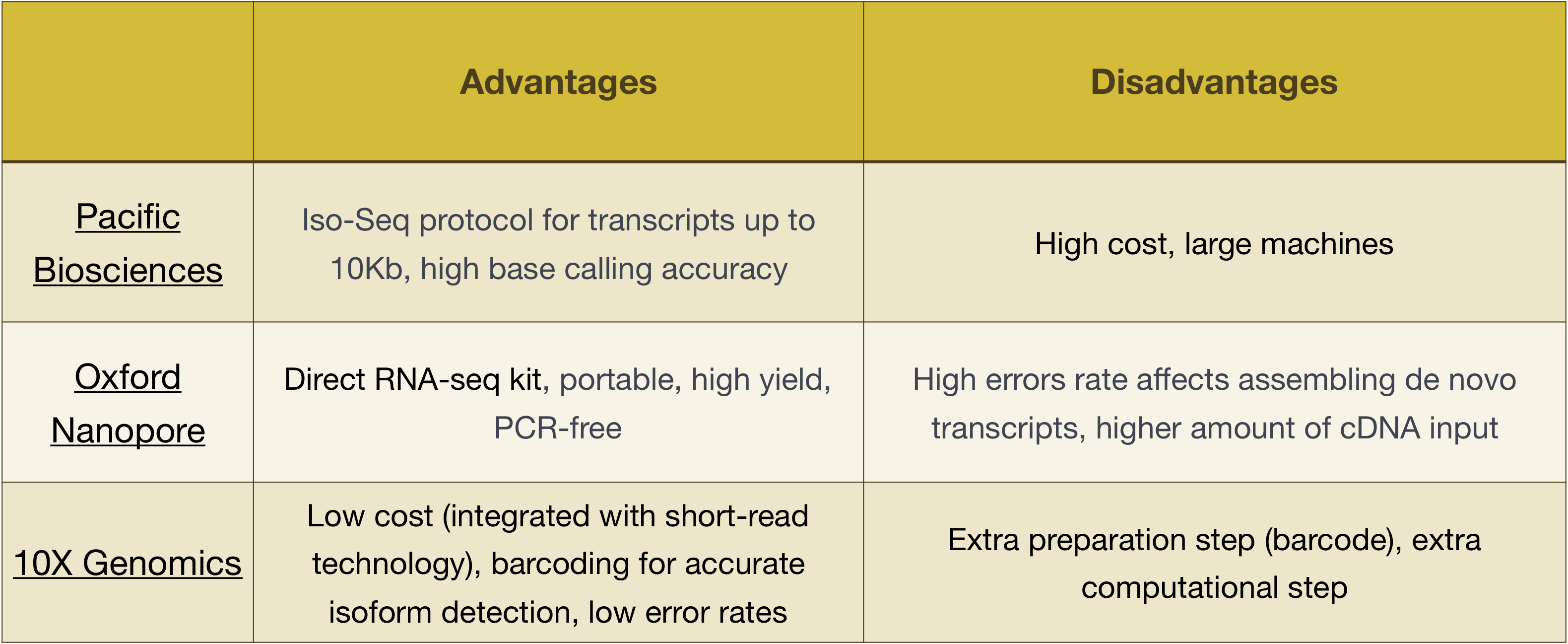
- Pacific Biosciences: http://www.pacb.com/
- Oxford Nanopore (MinION): https://nanoporetech.com/
- 10X Genomica: https://www.10xgenomics.com/
Avantajele și dezavantajele acestor tehnologii pot fi explorate în tabelul de mai jos:
Lasă un răspuns