
Genetischer Polymorphismus ist die Existenz alternativer DNA-Zustände, die die Variation der höheren Integrationsstufen des Organismus bestimmen. Es gibt verschiedene Arten von Genom-Veränderungen (Mutationen). Die am meisten untersuchten sind Nukleotidsubstitutionen in kodierenden und regulatorischen Regionen.
Definition
Genetischer Polymorphismus ist die Existenz mehrerer alternativer Zustände von DNA oder Allelen an einer definierten Position im Genom oder Ortposition des Gens auf dem Chromosom., In der Populationsgenetik eine Reihe von homologen Genen (Homologieklasse). Zwei Gene sind homolog, wenn sie mit der Meiose übereinstimmen. Plural: loci.. Diese Definition hat mehrere Aspekte:
(1) Erstens muss das Merkmal von den Chromosomen getragen werden und übertragbar sein.
(2) Dann müssen die Allele homolog seinzwei Chromosomen oder zwei Gene sollen homolog sein, wenn sie übereinstimmen und sich gegenseitig von der Meiose ausschließen. für ihre Position im Genom, die bestimmte Arten von Variationen ausschließt, die später diskutiert werden.,
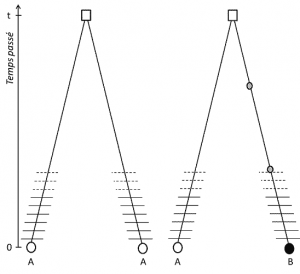
(3) Da das Merkmal jedoch übertragbar ist, impliziert die Positionshomologie auch, dass die Allele nach Abstammung homolog sind; Wenn sie unterschiedlich sind, ist es daher, dass (zumindest) eine Mutation in einer der Linien aufgetreten ist, die sie mit ihrem letzten gemeinsamen Vorfahren verbindet (Abbildung 1).
(4) Schließlich kann genetischer Polymorphismus auf der Skala der kleinsten Einheit, aus der DNA besteht, definiert werden: der Nukleotidstelle., Infolgedessen kann jede Nukleotidvariante den Polymorphismus der höheren Ebenen der biologischen Struktur – des Gens, Proteins und Phänotyps des Individuums – bestimmen, die zu Skalen werden, auf denen genetischer Polymorphismus beschrieben werden kann (Abbildung 2).
Nehmen Sie das Beispiel des ABO-Blutsystems, das einem antigenen motivenTeil des Antigens entspricht, das von einem Antikörper-oder Lymphozytenrezeptor erkannt wird. Auch bekannt als Epitop oder Antigene Determinante. Das gleiche Antigen kann mehrere Epitope (identisch oder unterschiedlich) aufweisen und somit eine unterschiedliche Immunantwort auslösen., beteiligt an der Kompatibilität während Bluttransfusionen. Dies ist ein Fall von genetischem Polymorphismus, der in menschlichen Populationen vorhanden ist und das Protein ABO Glycosyl Transferaseenzym umfasst, das die Übertragung von zuckerhaltigen Rückständen auf Proteine ermöglicht. Im Fall des ABO-Systems induzieren Glykosyltransferase A und B jeweils ein Individuum zur Gruppe A oder B. Wenn beide Glykosyltransferasen vorhanden sind, ist das Individuum der Gruppe AB. das hat drei Allele, A, B und O. Ein Individuum kann (AA), (AO); (BB), (BO); (OO) oder (AB) Genotypen haben., Sein Phänotyp wird dann sein,,;,; oder, wo wir sehen, dass A und B „dominieren“ O (dh ihr Ausdruck maskiert den von O), und dass sie untereinander ko-dominant sind (der heterozygote zwischen A und B hat einen erkennbaren Ausdruck,).
Ein Polymorphismus kann zunächst auf der Skala der DNA-Sequenz des für ein Protein kodierenden Locus beschrieben werden. Einige Polymorphismen sind „synonym“, d.h. sie verändern nicht die Aminosäuresequenz des Proteins; Sie sind oft die zahlreichsten Polymorphismen. Die anderen modifizieren Aminosäuren und werden als „Ersatz“ – Polymorphismen bezeichnet., Es gibt zwei Arten von Ersatzpolymorphismen der ABO-Glycosyltransferase: Polymorphismen, die die Aminosäure, aber nicht die antigene Einheit verändern (sie stören nicht den ABO-Phänotyp von Individuen); und andere Ersatzpolymorphismen, die das ABO-Phänotypset beobachtbarer Eigenschaften eines Individuums bestimmen..
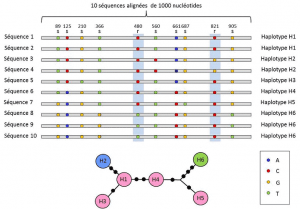
Zwischen den Nucleotid-und Allelebenen berücksichtigen genetische Analysen eine Zwischenebene der Beschreibung: die lineare Anordnung variabler Stellen am Ort. Diese Anordnungen, genannt haplotypegRuppe von Allelen verschiedener Loci, die sich auf demselben Chromosom befinden und normalerweise zusammen übertragen werden. Haplotype ist ein durch die Kontraktion des englischen Begriffs haploid Genotyp oder haploid Genotyp gebildet., Alle Gene, die sich auf demselben Chromosom befinden und deren Allele sich während der Meiose trennen, bilden einen Haplotyp. Diese Gene werden als „genetisch verknüpft“ bezeichnet und sind in der evolutionären Genetik nützlich, da sie es ermöglichen, die genealogischen Verbindungen zwischen den Allelen zu finden (Abbildung 2).
Nukleotidpolymorphismen existieren auch in intergenen Regionen (außerhalb codierender Regionen). Einige beeinflussen die Bereiche, in denen die Genexpression reguliert wird, und haben daher eine phänotypische Expression. Andere haben keine bekannte Wirkung und werden als „still“bezeichnet.,
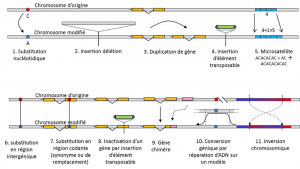
Neben Nukleotidsubstitutionen bezieht sich Polymorphismus für einen bestimmten Mutationstyp, Mikrosatelliten, auf Variationen in der Länge eines häufig nicht kodierenden DNA-Fragments (Abbildung 3) aufgrund einer Änderung der Anzahl von Wiederholungen in einer kurzen wiederholten Nukleotidsequenz, z. B. CACACACA oder TGTGTG.
Nicht jede genetische Variation fällt in die Definition des genetischen Polymorphismus, da sie die Positionshomologie von Alternativen erfordert (Abbildung 3)., Somit gibt es wiederholte Sequenzen, die durch Duplizieren eines Gens erhalten werden, für die zwei Wiederholungen pro Position nicht homolog sind. Oft können wir auch nicht über Homologie für transponierbare Elemente sprechen, da sie im Allgemeinen ihre Position während der Zellgenerationen ändern, sich vermehren und in das Genom eindringen können. Die Homologie zweier transponierbarer Elemente kann dann nicht definiert werden. Andererseits entsprechen zwei kodierende Sequenzen desselben Ortes, von denen eine intakt und die andere durch das Einfügen eines transposierbaren Elements inaktiviert ist, gut einer Homologiesituation., Eines Tages werden wir wahrscheinlich darüber nachdenken, über Polymorphismus für die Modifikationen sogenannter „epigenetischer“ Chromosomen zu sprechen, die manchmal über mehrere Zellgenerationen übertragen werden, somatischqualifiziert nicht reproduktive Zellen oder Soma. Eine somatische Mutation, die ein Gen einer somatischen Zelle beeinflusst, verschwindet mit dem Träger Individuum. oder germinalqualifiziert Gameten. Eine Keimlinienmutation kann auf ihre Nachkommen übertragen werden., wie Methylierung. Ihre Bedeutung für die Entwicklung der Populationen muss noch bewertet werden., Die Beschränkung des Begriffs des genetischen Polymorphismus auf bestimmte Variationskategorien – im Wesentlichen Nukleotidsubstitutionen – beruht auf dem großen Nutzen der letzteren bei der Erforschung der Geschichte natürlicher Populationen. Sie eignen sich zur mathematischen Modellierung der Evolution.
Das Wort Polymorphismus bedeutet „mehrere Formen“. Es steht im Gegensatz zum Monomorphismus, der auf das Fehlen von Variationen hinweist. Im Vokabular der Biologie wird diese Opposition seit langem vor der Genetik verwendet, um das Zusammenleben mehrerer Arten von Individuen in derselben Spezies zu signalisieren, z, Kastenpolymorphismus (Königin und Arbeiter) bei sozialen Insekten; saisonaler Polymorphismus (Fellveränderungen) bei einigen arktischen Säugetieren. Diese Fälle fallen nicht unter den genetischen Polymorphismus, ein neueres Konzept mit einer engeren und genaueren Bedeutung. Kontinuierliche Variationen (z. B. Größenvariationen) fallen ebenfalls nicht in die Kategorie des Polymorphismus, da sie keine unterschiedlichen Alternativen darstellen. Locus, die sich auf die Größe auswirken, fallen jedoch in diese Definition., Wir werden im Folgenden die am meisten untersuchten genetischen Variationen diskutieren, da sie für die Evolution am wichtigsten sind, Nukleotidsubstitutionen, und dann werden wir die Variation von Phänotypen diskutieren.
Messung
Es genügt nicht zu sagen, dass ein Ort mehr oder weniger variabel ist, ohne diesem Urteil eine quantitative Schätzung zu geben. Ein Forscher kann die Variation auf verschiedenen Skalen untersuchen. Wenn er sich nur für die Allele eines Proteins interessiert, misst er die Allelvielfalt, symbolisiert durch „H“. Wenn er sich für die DNA-Vielfalt interessiert, misst er die Nukleotid-Vielfalt, symbolisiert durch „π“.,
Die Allelvielfalt H ist definiert als die Wahrscheinlichkeit, zwei verschiedene Allele während zwei Probenahmen mit Ersatz zu zeichnendurchlaufen einer aufeinanderfolgenden Auslosung mit Lieferung von p-Token in einer Urne, die n Token enthält, bedeutet, ein erstes Token zu nehmen, seinen Wert zu lesen, es wieder in die Urne zu setzen, ein zweites Token zu nehmen, seinen Wert zu lesen, es wieder in die Urne zu setzen usw., bis zum pth-Token. Dies bedeutet, dass Sie p Objekte unter n mit Wiederholung auswählen (Sie können dasselbe Objekt mehrmals auswählen) und in der Reihenfolge (die Reihenfolge, in der Sie die Objekte auswählen, ist wichtig)., Die Anzahl der aufeinanderfolgenden Ziehungen mit Token unter n ist: n × n × n × … × n = np.. Wenn wir pi die Frequenz des Allels des Ranges i nennen, zeigen wir, dass die Wahrscheinlichkeit, zweimal dasselbe Allel zu sammeln, F = Σ pi2 ist. Die allelische Vielfalt der Probe wird dann deren Ergänzung zu 1, d.h.:
H = 1 – Σ pi2 (1)
Dies gilt sowohl für protein Allele und haplotypen. Dies nennt man haplotypische Vielfalt. Es kann auch als Heterozygotie bezeichnet werden, da es bei einem diploiden Ort die erwartete Häufigkeit von Heterozygoten ergibt.,
Die Nukleotidvielfalt π ist das Äquivalent von H auf der Skala jedes Nukleotids. Es wird als Durchschnitt der Anzahl der Nukleotiddifferenzen zwischen Sequenzen in einer Probe berechnet, die zwei mal zwei (δij) entnommen wurden, geteilt durch die Länge des DNA-Fragments in die Anzahl der Nukleotide (L).
π = Mittelwert (δij)/L (2) (siehe Hinweis )
Dieser Wert unterscheidet sich zwischen den Arten. In den kodierenden Teilen des menschlichen Genoms unterscheiden sich zwei zufällig aus der Population ausgewählte Chromosomen im Durchschnitt um ein Nukleotid pro Tausend., In der Fruchtfliege (Drosophila melanogaster) ist dieser Unterschied etwa einer von hundert. Die Fliege ist somit zehnmal variabler als der Mensch. Wenn diese Werte mit der Größe der kodierenden Regionen (etwa 15.500 Gene in Drosophila, mindestens 22.000 beim Menschen) oder sogar mit Genomen (140 Millionen Basenpaare pro haploidem Genom bei Fliegen, etwa 3.200 Millionen oder zwanzigmal mehr beim Menschen) zusammenhängen, ist die Anzahl der polymorphen Stellen astronomisch und führt dazu, dass niemand von der sexualisierten Generation in irgendeiner Art geboren wird, war und ist genetisch identisch mit einer anderen Vergangenheit, Gegenwart oder Zukunft., Die Informationskraft des DNA-Polymorphismus ist immens. In der Forensik können Detektive jeden Verdächtigen aus sechzehn Mikrosatellitenlokalen identifizieren.
Geschichte
Der Begriff Variation steht im Titel der ersten beiden Kapitel von Darwins Buch The Origin of Species (1859). Darwin führte diesen Begriff in die Naturwissenschaften zu einer Zeit ein, als die Gesetze der biologischen Vererbung ein Rätsel blieben, und widmete ihm ein weiteres wichtiges Buch, Variation in Tieren und Pflanzen (1868)., In der Überzeugung, dass es bei der Evolution um kleine Variationen ging, die wenig Unterschied für die Anpassung von Organismen an ihre Lebensbedingungen machten, legte er großen Wert auf kleine quantitative Variationen, was seine Nachfolger (insbesondere Karl Pearson) dazu veranlasste, die biometryScience of Life Measurement zu gründen. Bezieht sich im weitesten Sinne auf die quantitative Untersuchung von Lebewesen.. Aber die Wiederentdeckung von Mendels Gesetzen im Jahr 1900 verlagerte das Interesse auf diskontinuierliche Variationen.
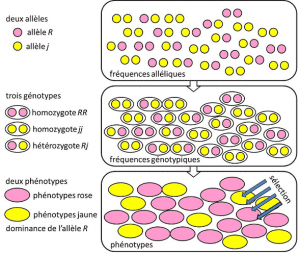
Von 1908 bis 1930 war Populationsgenetik im Wesentlichen eine theoretische Disziplin, die Darwinismus mit mendelschen Vererbung in Einklang bringen wollte, basierend auf der Übertragung eines einzelnen Gens unter einem dominanten, rezessiven oder geschlechtschromosomengebundenen Modus X (oder Y). Bezieht sich auf ein genetisches Merkmal mit einfachem Determinismus, durch ein Paar oder eine kleine Anzahl von Paaren von Genen., und wo Wahrscheinlichkeiten eine große Rolle spielten. Genetik ist kontraintuitiv., Es sagt voraus, dass es in seinem Nachkommen keine Reproduktion des Elternteils gibt, da dessen Genotyp aus der Verletzung der Trennung der Allele vor der Verschmelzung der beiden elterlichen Halbgenome resultiert. Es wurde erkannt, dass dies die Allelfrequenzen sindfrequenz, bei der das Allel einer Variante in einer Population gefunden wird. Ausgedrückt als Anteil oder Prozentsatz. Die Summe der Allelfrequenzen aller Allele eines Gens in einer Population ist daher per Definition gleich 1. In der Populationsgenetik repräsentieren Allelfrequenzen die genetische Vielfalt auf Populations-oder Artenebene., die zwischen Generationen übertragen werden, nicht Genotypen oder Phänotypen. Diese Frequenzen sind von Generation zu Generation mehr oder weniger stabil und erzeugen jenseits der multiplen Verschränkung von Filiationen zwischen Individuen die gleichen genotypischen Häufigkeitengenetische Struktur der Bevölkerung. Bestimmt aus den Allelfrequenzen (Abbildung 4). Die Verteilung der Populationsgenotypen, die mit Varianz verbunden ist, ist daher der einzige vorhersehbare Faktor., Um 1930 halfen die drei Theoretiker Ronald Fisher, JBS Haldane und Sewall Wright, den Begriff der mendelschen Bevölkerungbevölkerung, deren Vererbung Mendels Gesetzen gehorchte. . In diesem Zusammenhang assoziiert die Evolution notwendigerweise drei strukturelle Ebenen: das Gen, das Individuum und die Bevölkerung. Diese Drei-Wege-Assoziation kann durch die folgende Formel zusammengefasst werden: Populationsentwicklung ist eine Änderung der Allelfrequenz (Genskala, aber auch Gruppenskala), eine Änderung, die von der Sortierung der Phänotypen nach Selektion abhängt (individuelle Skala)., Die empirische Populationsgenetikforschung entwickelte sich dann, war jedoch lange Zeit durch unsere Unkenntnis der Funktionsweise von Chromosomen begrenzt (die Struktur der DNA wurde 1953 verstanden und ihre Sequenzierung begann 1977) und musste auf das Studium der wenigen sichtbaren Polymorphismen zurückgreifen, wie die Färbung von Marienkäfer-Elytra oder die Schale von Schnecken (siehe Fokus Die große Schneckendebatte). Es gab viele Debatten unter Genetikern darüber, ob natürliche Populationen im Allgemeinen polymorph oder monomorph waren und ob Polymorphismus an sich vorteilhaft war oder nicht., Die Untersuchung der Variation der makromolekularen Skala musste bis 1966 (für Proteine) und 1983 (für DNA) warten, bevor diese Fragen beantwortet wurden.
Quantitative Variationen und Erblichkeit
Wenn wir uns umsehen, sehen wir physische Unterschiede zwischen Wesen, einige komplex wie Gesichtszüge, andere leicht zu messen wie Gewicht oder Größe. Der gesunde Menschenverstand legt nahe, dass sie teilweise übertragbar sind, wenn auch auf eine schwer zu spezifizierende Weise. Diese Merkmale sind oft polygenunter der Abhängigkeit vieler Gene. Wir sprechen über polygene Vererbung. Diabetes ist eine polygene Erkrankung.,, was bedeutet, dass sie von vielen Locuses beeinflusst werden. Die Entwicklungsgenetik und ihre Anwendung auf den Vergleich von Arten (evo-devo, für „Evolution der Entwicklung“) entwirren das Netz komplexer Interaktionen, die dazu führen, dass sich die Zellen des Körpers, die alle identisches genetisches Gepäck haben, während der Entwicklung durch Epigenese differenzieren, um verschiedene Gewebe zu erhalten. Es ist immer noch unklar, wie komplexe Charaktere aufgebaut sind. Messbare Phänotypen können anhand von Biometrie anhand der Eigenschaften statistischer Verteilungen untersucht werden., Ein Wert mit nützlichen Eigenschaften ist die Varianzin einer Stichprobe von Individuen, in denen ein bestimmtes Merkmal gemessen wurde, ist die Varianz die Differenz zwischen dem Mittelwert des Wertquadrats und dem Quadrat des Mittelwerts der Werte. Diese Maßnahme, die immer positiv ist, zeigt die Streuung von Individuen an. . Wenn mehrere unabhängige Ursachen die Variation einer Linie bestimmen, sind ihre Varianzen additiv und ihre Summe ergibt die Varianz der Linie. Wenn sie nicht unabhängig sind, wird die Summe der Kovarianzen zur Summe der Varianzen addiert., Die Gesamtvarianz Vt eines phänotypischen Merkmals ist so, dass :
Vt = Vga+Vgd+Vgd+Vgi+Ve
Vga, additive genetische Varianz, die Summe der Varianzen ist, die auf jeden Ort unabhängig von den anderen zurückzuführen sind; Vgd, dominante genetische Varianz, ergibt sich aus der Wechselwirkung der beiden Allele desselben Ortes; Vgi, Interaktionsvarianz zwischen Orten, ergibt sich aus der Tatsache, gleiches Individuum interagieren; Ve, Varianz der Umwelt, wird hier unabhängig von genetischer Varianz angenommen.,
Die genetische Varianz der Dominanz ist zwei Kindern derselben Eltern gemeinsam, weil sie Dominanzeffekte teilen, wenn sie an einem bestimmten Ort die gleichen Allele von beiden Elternteilen erhalten haben, was ihnen an einem von vier Orten passiert. Sie teilen diese Effekte nicht mit ihren Eltern, daher sind sie einander „ähnlicher“ als ihre Eltern, obwohl sie so viele Gene miteinander teilen. Sicherlich kann ein Elternteil zufällig die gleichen zwei Allele wie eines seiner Kinder an einem bestimmten Ort erhalten haben., Diese Chance hängt von der Häufigkeit von Allelen in der Bevölkerung ab. Wir sehen, dass der Grad der Ähnlichkeit zwischen einem Kind und seinem Elternteil für ein bestimmtes Merkmal ein öffentliches Eigentum ist.
Im Allgemeinen können alle Komponenten der genetischen Varianz (Vga+Vgd+Vgi) zwischen den Populationen variieren. In Zuchtlinien ist Inzucht ein häufiger Fall, der die genetische Varianz erschöpft: infolgedessen kann ein Merkmal in zwei Populationen den gleichen Durchschnitt haben, aber eine andere genetische Variation aufweisen. Die Fähigkeit eines Züchters, ein Merkmal auszuwählen, ist daher ein Merkmal der Linie, nicht des Merkmals.,
Die Beziehung zwischen Eltern und Kindern wird an der Erblichkeit h2 gemessen. Es ist definiert als das Verhältnis der Additiven genetischen Varianz zur gesamtvarianz :
h2 = Vga/Vt

(2) Korrelation von 0, wobei die Größe der Kinder unabhängig von der der Eltern ist. Ein wertvoller durchschnittlicher Elternteil hat ein Kind von gleichgültigem Wert innerhalb der Verteilung von Kindern hervorgebracht. Die Koordinaten der Punkte befinden sich auf einem Kreis, die Erwartung ihres Wertes befindet sich auf einer Linie parallel zur Achse der Eltern (d)., (3) Zwischen Eltern-Kind-Korrelation zwischen 0 und 1: Die Koordinaten der Punkte befinden sich auf einer Ellipse. Die Erwartung ihres Wertes befindet sich nicht mehr auf der winkelhalbierenden der Koordinatenachsen (die auch die Hauptachse der Verteilungsellipse ist), sondern auf der Regressionslinie r. Ein durchschnittlicher Elternteil des Wertes hat ein Kind des Wertes b (zwischen 0 und a) mit einer gewissen Varianz erzeugt. Es ist anzumerken, dass der Fall (3) zwischen den Fällen (1) und (2)
liegt Es wird unter verschiedenen Annahmen in Abbildung 5 gezeigt., Erblichkeit ist die Fähigkeit zu bestimmen, ob ein Merkmal ausgewählt werden kann. Abbildung 5-3 zeigt, dass ein Züchter innerhalb einer Übergangsgeneration in der Lage sein wird, den Durchschnitt eines Zeichens vom Wert 0 auf den Wert b zu verschieben, wenn er Individuen mit dem Wert a auswählt. Wir können zeigen, dass b = h2a. Um ein Merkmal auszuwählen, ist es nützlich, das Erbschaftsverhältnis zu erhöhen, indem die Umwelt standardisiert wird (wodurch die Umweltvarianz verringert wird) und indem man sich in die Umweltbedingungen einfügt, die das Erscheinungsbild des Merkmals begünstigen., Der Genetiker Waddington hat gezeigt, dass eine Veränderung der Umwelt in der Natur neue Merkmale aufdecken kann, die dann während der Evolution ausgewählt werden.
Referenzen und Hinweise
Titelbild. Snail shells Cepaea nemoralis & Cepaea hortensis. André Künzelmann, UFZ.,
Diese Formel kann konventioneller geschrieben werden als: \pi =\frac{n (n-1))}{2L}\sum _{i=1}^{n-1}\sum_{j=i+1}^{n}\delta ij
Als Stichprobe von Personen, bei denen das untersuchte Merkmal gemessen wurde, kann die Varianz als Differenz zwischen dem Mittelwert des Wertquadrats und dem Quadrat des Mittelwerts der Werte definiert werden. Diese Maßnahme, die immer positiv ist, zeigt die Streuung von Individuen an.
Dies ist Erblichkeit im engeren Sinne des Begriffs. Erblichkeit im weiten Sinne das Verhältnis der Summe des genetischen Abweichungen zu Gesamtabweichung., Diese Formel gibt Erblichkeit mit beiden Elternteilen (dem „durchschnittlichen Elternteil“); Die Schätzung mit nur einem Elternteil würde h2/2 ergeben.
Die Umwelt-Enzyklopädie, die Umwelt, die von der Association des Encyclopédies de l ‚Environnement et de l‘ Énergie (www.a3e.fr), vertraglich verbunden mit der Universität Grenoble Alpes und Grenoble INP, und gesponsert von der französischen Akademie der Wissenschaften.
Um diesen Artikel zu zitieren: VEUILLE Michel (2021), Genetischer Polymorphismus und Variation, Enzyklopädie der Umwelt, url : https://www.encyclopedie-environnement.org/en/life/genetic-polymorphism-and-variation/.,
Die Artikel in der Encyclopedia of the Environment werden unter den Bedingungen der Creative Commons BY-NC-SA-Lizenz zur Verfügung gestellt, die die Reproduktion autorisiert, vorbehaltlich: unter Berufung auf die Quelle, keine kommerzielle Nutzung von ihnen, identische Anfangsbedingungen teilen, bei jeder Wiederverwendung oder Verteilung die Erwähnung dieser Creative Commons BY-NC-SA-Lizenz reproduzieren.
Schreibe einen Kommentar